4 Oracle Machine Learning for Rでのモデルの構築
OML4Rには、回帰モデル、ニューラル・ネットワーク・モデルおよびOracle Machine Learning for SQLアルゴリズムに基づいたモデルを構築するための関数が用意されています。
この章では、次の項目について説明します。
- Oracle Machine Learning for Rモデルの構築
OML4RのパッケージOREmodelsには、ore.frameオブジェクトを使用して拡張分析データ・モデルを作成できる関数が含まれています。 - Oracle Machine Learning for SQLのモデルの構築
Oracle Machine Learning for RのOREdmパッケージの関数を使用して、RでOracle Machine Learning for SQLのモデルを構築します。 - モデルの交差検定
交差検定とはモデル改善手法の1つであり、使用可能なデータから繰り返しサンプリングを行って複数のモデルを構築およびテストすることにより、1回の学習/試験実験に伴う制約を回避します。
4.1 Oracle Machine Learning for Rのモデルの構築
- OREmodels関数について
OREmodelsパッケージには、ore.frameオブジェクトを使用して機械学習モデルを構築できる関数が含まれています。 - サンプルのlongleyデータセットについて
ほとんどの線形回帰およびore.neuralの例では、Rによって提供されるlongleyデータセットを使用します。 - 線形回帰モデルの構築
ore.lm関数およびore.stepwise関数は、最小二乗回帰およびステップワイズ最小二乗回帰をそれぞれore.frameオブジェクトに表されたデータで実行します。 - 一般化線形モデルの構築
ore.glm関数は、ore.frameオブジェクト内のデータに対して一般化線形モデルを適合します。 - ニューラル・ネットワーク・モデルの構築
ニューラル・ネットワーク・モデルを使用すると、入力と出力との間の複雑な非リニアの関係を取得すること、つまりデータのパターンを見つけることができます。 - ランダム・フォレスト・モデルの構築
ore.randomForest関数は、ore.frameオブジェクト内のデータを分類するためのアンサンブル学習手法を提供します。
4.1.1 OREmodels関数について
OREmodelsパッケージには、ore.frameオブジェクトを使用して機械学習モデルを構築できる関数が含まれています。
OREmodelsの関数を次に示します。
表4-1 OREmodelsパッケージの関数
| 関数 | 説明 |
|---|---|
|
|
|
|
|
|
|
|
|
ore.randomForest |
ore.frame内のデータに基づいてランダム・フォレスト分類モデルを並行して作成します。
|
|
|
|
ノート:
Rの用語では、「モデルを適合する」という表現は、多くの場合「モデルを構築する」と同じ意味で使用されます。このドキュメントおよびOracle Machine Learning for Rの関数のオンライン・ヘルプでは、この表現は同じ意味で使用されます。
関数ore.glm、ore.lmおよびore.stepwiseには次の利点があります。
-
アルゴリズムはアウトオブコアQR Factorizationを使用した正確な解決方法を提供します。QR Factorizationは、行列を直交行列と三角行列に分解します。
QRは、困難とされるランクのない適合モデルに使用されるアルゴリズムです。
-
メモリーに収まらないデータ、つまりアウトオブコアのデータを処理できます。QRはマトリクスを、メモリーに収めるものとディスクに保存するものの、2つに分解します。
ore.glm、ore.lmおよびore.stepwiseの各関数は、10億行を超えるデータセットを解決できます。 -
ore.stepwise関数によって、前方、後方およびステップワイズによるモデル選択の手法を高速に実装できます。
ore.neural関数には次の利点があります。
-
ニューラル・ネットワークのスケーラビリティの高い実装であり、10億行のデータセット上のモデルであってもわずか数分で構築できます。
ore.neural関数は、小中規模のデータセットに対してはインメモリー、大規模入力に対しては分散(アウトオブコア)の2つのモードで実行できます。 -
ore.neuralでは多くの種類の活性化関数がサポートされているため、活性化関数をニューロン上で層ごとに指定できます。 -
0 (ゼロ)を含む任意の数の非表示層からなるニューラル・ネットワーク・トポロジを指定できます。
4.1.2 サンプルのlongleyデータセットについて
ほとんどの線形回帰およびore.neuralの例では、Rによって提供されるlongleyデータセットを使用します。
longleyデータセットは小さいマクロ経済データセットで、共線回帰の既知の例を提供し、16年にわたり毎年観察された7つの経済変数で構成されます。
例4-1 longleyデータセットの値の表示
この例では、longleyデータセットを一時データベース表にプッシュしますが、これにはプロキシore.frameオブジェクトのlongley_ofがあり、longley_ofの最初の6行を表示します。
longley_of <- ore.push(longley) head(longley_of)
この例のリスト
R> longley_of <- ore.push(longley)
R> dim(longley_of)[1] 16 7
R> head(longley_of)
GNP.deflator GNP Unemployed Armed.Forces Population Year Employed
1947 83.0 234.289 235.6 159.0 107.608 1947 60.323
1948 88.5 259.426 232.5 145.6 108.632 1948 61.122
1949 88.2 258.054 368.2 161.6 109.773 1949 60.171
1950 89.5 284.599 335.1 165.0 110.929 1950 61.187
1951 96.2 328.975 209.9 309.9 112.075 1951 63.221
1952 98.1 346.999 193.2 359.4 113.270 1952 63.6394.1.3 線形回帰モデルの構築
ore.lm関数およびore.stepwise関数は、最小二乗回帰およびステップワイズ最小二乗回帰をそれぞれore.frameオブジェクトに表されたデータで実行します。
モデル適合は、埋込みRマップ/リデュース処理を使用して生成されますが、このときマップ処理によって、評価される係数の数に応じてQR分解またはマトリクスのクロス積のいずれかが作成されます。基礎となるモデル・マトリクスは、モデルのスパーシティに応じてmodel.matrixオブジェクトまたはsparse.model.matrixオブジェクトのいずれかを使用して作成されます。モデルの係数が推定されると、データの別のパスが実行されて、モデル水準統計が推定されます。
前方、後方またはステップワイズ検索が実行されるとき、XtXおよびXtyマトリクスがサブセット化され、XtXサブセット・マトリクスのコレスキー分解を使用して生成された係数推定に基づくF検定のp値が生成されます。
モデルに共線的な項がある場合、関数ore.lmおよびore.stepwiseは一連の共線的な項に対する係数値を推定しません。ore.stepwiseでは、共線的な項セットは、手順全体から除外されます。
ore.lmおよびore.stepwiseの詳細は、help(ore.lm)を呼び出してください。
例4-2 ore.lmの使用方法
この例では、longleyデータセットを一時データベース表にプッシュしますが、これにはプロキシore.frameオブジェクトのlongley_ofが含まれています。この例では、ore.lmを使用して線形回帰モデルを構築します。
longley_of <- ore.push(longley) # Fit full model oreFit1 <- ore.lm(Employed ~ ., data = longley_of) class(oreFit1) summary(oreFit1)
この例のリスト
R> longley_of <- ore.push(longley)
R> # Fit full model
R> oreFit1 <- ore.lm(Employed ~ ., data = longley_of)
R> class(oreFit1)
[1] "ore.lm" "ore.model" "lm"
R> summary(oreFit1)
Call:
ore.lm(formula = Employed ~ ., data = longley_of)
Residuals:
Min 1Q Median 3Q Max
-0.41011 -0.15767 -0.02816 0.10155 0.45539
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -3.482e+03 8.904e+02 -3.911 0.003560 **
GNP.deflator 1.506e-02 8.492e-02 0.177 0.863141
GNP -3.582e-02 3.349e-02 -1.070 0.312681
Unemployed -2.020e-02 4.884e-03 -4.136 0.002535 **
Armed.Forces -1.033e-02 2.143e-03 -4.822 0.000944 ***
Population -5.110e-02 2.261e-01 -0.226 0.826212
Year 1.829e+00 4.555e-01 4.016 0.003037 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.3049 on 9 degrees of freedom
Multiple R-squared: 0.9955, Adjusted R-squared: 0.9925
F-statistic: 330.3 on 6 and 9 DF, p-value: 4.984e-10
例4-3 ore.stepwise関数の使用方法
この例では、longleyデータセットを一時データベース表にプッシュしますが、これにはプロキシore.frameオブジェクトのlongley_ofが含まれています。この例では、ore.stepwise関数を使用して線形回帰モデルを構築します。
longley_of <- ore.push(longley)
# Two stepwise alternatives
oreStep1 <-
ore.stepwise(Employed ~ .^2, data = longley_of, add.p = 0.1, drop.p = 0.1)
oreStep2 <-
step(ore.lm(Employed ~ 1, data = longley_of),
scope = terms(Employed ~ .^2, data = longley_of))この例のリスト
R> longley_of <- ore.push(longley)
R> # Two stepwise alternatives
R> oreStep1 <-
+ ore.stepwise(Employed ~ .^2, data = longley_of, add.p = 0.1, drop.p = 0.1)
R> oreStep2 <-
+ step(ore.lm(Employed ~ 1, data = longley_of),
+ scope = terms(Employed ~ .^2, data = longley_of))
Start: AIC=41.17
Employed ~ 1
Df Sum of Sq RSS AIC
+ GNP 1 178.973 6.036 -11.597
+ Year 1 174.552 10.457 -2.806
+ GNP.deflator 1 174.397 10.611 -2.571
+ Population 1 170.643 14.366 2.276
+ Unemployed 1 46.716 138.293 38.509
+ Armed.Forces 1 38.691 146.318 39.411
<none> 185.009 41.165
Step: AIC=-11.6
Employed ~ GNP
Df Sum of Sq RSS AIC
+ Unemployed 1 2.457 3.579 -17.960
+ Population 1 2.162 3.874 -16.691
+ Year 1 1.125 4.911 -12.898
<none> 6.036 -11.597
+ GNP.deflator 1 0.212 5.824 -10.169
+ Armed.Forces 1 0.077 5.959 -9.802
- GNP 1 178.973 185.009 41.165
... The rest of the output is not shown.4.1.4 一般化線形モデルの構築
ore.glm関数は、ore.frameオブジェクト内のデータに対して一般化線形モデルを適合します。
この関数は、Fisherスコアリングの繰返し加重最小二乗(IRLS)アルゴリズムを使用します。適切ではない係数推定の選択を防ぐ必要がある従来のステップのかわりに、ore.glmは行検索を使用して、繰返しごとに新しい係数推定を選択し、式(1 - alpha) * old + alpha * suggestedを使用して現在の係数推定から開始し、Fisherスコアリングで提案される推定に移動します(ここでalphaは[0, 2]です)。interp制御引数がTRUEの場合、逸脱度は3次スプライン補間によって概算されます。FALSEの場合、逸脱度はフォローアップ・データ・スキャンを使用して計算されます。
各繰返しは、2つまたは3つの埋込みRマップ/リデュース操作で構成されますが、それは、IRLS操作、初期行検索操作およびinterp = FALSEの場合は、オプションのフォローアップ行検索操作です。ore.lmを使用すると、IRLSマップ操作によって、QR分解(update = "qr"の場合)、クロス積(model.matrixのupdate = "crossprod"の場合)またはsparse.model.matrix (引数sparse = TRUEの場合)が作成され、IRLSリデュース操作ブロックは、それらのQR分解またはクロス積行列を更新します。アルゴリズムが収束するか、最大繰返し数に到達したら、最後の埋込みRマップ/リデュース操作が使用され、完全なモデル水準統計が生成されます。
ore.glm関数は、ore.glmオブジェクトを返します。
ore.glm関数の引数の詳細は、help(ore.glm)をコールしてください。
一般化線形モデルの設定
次の表に、一般化線形モデルに適用される設定を示します。
表4-2 一般化線形モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
信頼区間の信頼水準。 デフォルトの信頼水準は |
|
|
|
特徴生成が二次式または三次式のどちらであるかを示します。 特徴の生成を有効にすると、アルゴリズムでは、最適な特徴生成方式がデータに基づいて自動的に選択されます。 |
|
|
|
GLMに対して特徴生成を有効にするかどうかの指定。デフォルトでは、特徴生成は有効化されていません。 ノート: 特徴生成は、特徴選択が有効な場合にのみ有効です。 |
|
|
|
モデルに特徴を追加する際の、特徴選択のペナルティ基準。 特徴選択を有効にすると、アルゴリズムでは、ペナルティ基準がデータに基づいて自動的に選択されます。 |
|
|
|
GLMに対して特徴選択を有効にするかどうかの指定。 デフォルトでは、特徴選択は無効です。 |
|
|
|
特徴選択を有効にすると、この設定では、最終モデルで選択可能な特徴の最大数が指定されます。 デフォルトでは、十分なメモリーを確保するために、アルゴリズムによって特徴の数が制限されます。 |
GLMS_PRUNE_MODEL |
|
最終モデルでの特徴のプルーニングを有効また無効にします。プルーニングは、線形回帰の場合はt検定統計に、ロジスティック回帰の場合はWald検定統計に基づきます。すべての特徴が全データに対して統計的な意味付けを持つまで、これらの特徴はループ処理でプルーニングされます。 特徴選択を有効にすると、アルゴリズムでは、プルーニングがデータに基づいて自動的に実行されます。 |
|
|
target_value |
バイナリ・ロジスティック回帰モデルで参照クラスとして使用されるターゲット値。非参照クラスに対しては確率が生成されます。 デフォルトでは、参照クラスの場合、最も普及率の高い値(ほとんどのケース)がアルゴリズムによって選択されます。 |
GLMS_RIDGE_REGRESSION |
|
リッジ回帰を有効または無効にします。リッジは、回帰と分類の両方のマイニング機能に適用されます。 リッジを有効にすると、 ノート: リッジは、特徴選択が指定されていない場合または明示的に無効にされている場合にのみ有効です。リッジ回帰と特徴選択を両方とも明示的に有効にすると、例外が発生します。 |
|
|
|
リッジ・パラメータ値。この設定は、アルゴリズムでリッジ回帰の使用が構成されている場合にのみ使用されます。 リッジ回帰がアルゴリズムによって内部的に有効になっている場合、リッジ・パラメータはアルゴリズムによって決定されます。 |
|
|
|
行診断を有効または無効にします。 |
|
|
範囲は( |
GLMアルゴリズムの収束許容値の設定 デフォルト値は、システムによって決定されます。 |
|
|
正の整数 |
GLMアルゴリズムの最大反復数。デフォルト値は、システムによって決定されます。 |
|
|
0または正の整数
|
SGDソルバーで使用されるバッチ内の行数。このパラメータの値によって、SGDソルバーのバッチのサイズが設定されます。0を入力すると、データ駆動のバッチ・サイズの推定値がトリガーされます。 デフォルトは |
|
|
|
この設定により、ユーザーがGLMソルバーを選択できます。 |
|
|
|
この設定により、ユーザーはスパース・ソルバーを使用できます(使用可能な場合)。デフォルト値は |
例4-4 ore.glm関数の使用方法
この例では、rpartパッケージをロードした後、プロキシore.frameオブジェクトのKYPHOSISが含まれている一時データベース表にkyphosisデータセットをプッシュします。この例では、ore.glm関数およびglm関数を使用して一般化線形モデルをそれぞれ構築し、このモデルでsummary関数をコールします。
# Load the rpart library to get the kyphosis and solder data sets. library(rpart) # Logistic regression KYPHOSIS <- ore.push(kyphosis) kyphFit1 <- ore.glm(Kyphosis ~ ., data = KYPHOSIS, family = binomial()) kyphFit2 <- glm(Kyphosis ~ ., data = kyphosis, family = binomial()) summary(kyphFit1) summary(kyphFit2)例4-4のリスト
R> # Load the rpart library to get the kyphosis and solder data sets.
R> library(rpart)
R> # Logistic regression
R> KYPHOSIS <- ore.push(kyphosis)
R> kyphFit1 <- ore.glm(Kyphosis ~ ., data = KYPHOSIS, family = binomial())
R> kyphFit2 <- glm(Kyphosis ~ ., data = kyphosis, family = binomial())
R> summary(kyphFit1)
Call:
ore.glm(formula = Kyphosis ~ ., data = KYPHOSIS, family = binomial())
Deviance Residuals:
Min 1Q Median 3Q Max
-2.3124 -0.5484 -0.3632 -0.1659 2.1613
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -2.036934 1.449622 -1.405 0.15998
Age 0.010930 0.006447 1.696 0.08997 .
Number 0.410601 0.224870 1.826 0.06786 .
Start -0.206510 0.067700 -3.050 0.00229 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 83.234 on 80 degrees of freedom
Residual deviance: 61.380 on 77 degrees of freedom
AIC: 69.38
Number of Fisher Scoring iterations: 4
R> summary(kyphFit2)
Call:
glm(formula = Kyphosis ~ ., family = binomial(), data = kyphosis)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.3124 -0.5484 -0.3632 -0.1659 2.1613
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -2.036934 1.449575 -1.405 0.15996
Age 0.010930 0.006446 1.696 0.08996 .
Number 0.410601 0.224861 1.826 0.06785 .
Start -0.206510 0.067699 -3.050 0.00229 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 83.234 on 80 degrees of freedom
Residual deviance: 61.380 on 77 degrees of freedom
AIC: 69.38
Number of Fisher Scoring iterations: 5
# Poisson regression
R> SOLDER <- ore.push(solder)
R> solFit1 <- ore.glm(skips ~ ., data = SOLDER, family = poisson())
R> solFit2 <- glm(skips ~ ., data = solder, family = poisson())
R> summary(solFit1)
Call:
ore.glm(formula = skips ~ ., data = SOLDER, family = poisson())
Deviance Residuals:
Min 1Q Median 3Q Max
-3.4105 -1.0897 -0.4408 0.6406 3.7927
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -1.25506 0.10069 -12.465 < 2e-16 ***
OpeningM 0.25851 0.06656 3.884 0.000103 ***
OpeningS 1.89349 0.05363 35.305 < 2e-16 ***
SolderThin 1.09973 0.03864 28.465 < 2e-16 ***
MaskA3 0.42819 0.07547 5.674 1.40e-08 ***
MaskB3 1.20225 0.06697 17.953 < 2e-16 ***
MaskB6 1.86648 0.06310 29.580 < 2e-16 ***
PadTypeD6 -0.36865 0.07138 -5.164 2.41e-07 ***
PadTypeD7 -0.09844 0.06620 -1.487 0.137001
PadTypeL4 0.26236 0.06071 4.321 1.55e-05 ***
PadTypeL6 -0.66845 0.07841 -8.525 < 2e-16 ***
PadTypeL7 -0.49021 0.07406 -6.619 3.61e-11 ***
PadTypeL8 -0.27115 0.06939 -3.907 9.33e-05 ***
PadTypeL9 -0.63645 0.07759 -8.203 2.35e-16 ***
PadTypeW4 -0.11000 0.06640 -1.657 0.097591 .
PadTypeW9 -1.43759 0.10419 -13.798 < 2e-16 ***
Panel 0.11818 0.02056 5.749 8.97e-09 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 6855.7 on 719 degrees of freedom
Residual deviance: 1165.4 on 703 degrees of freedom
AIC: 2781.6
Number of Fisher Scoring iterations: 44.1.5 ニューラル・ネットワーク・モデルの構築
ニューラル・ネットワーク・モデルを使用すると、入力と出力との間の複雑な非リニアの関係を取得すること、つまりデータのパターンを見つけることができます。
ニューラル・ネットワーク・モデルの設定
次の表に、ニューラル・ネットワーク・モデルに適用される設定を示します。
表4-3 ニューラル・ネットワーク・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
NNET_HIDDEN_LAYERS |
負でない整数 |
非表示レイヤーの数によってトポロジを定義します。 デフォルト値は |
|
|
正の整数のリスト |
レイヤー当たりのノード数によってトポロジを定義します。様々なレイヤーに様々な数のノードを含めることができます。 この値は負数ではない整数にして、カンマで区切る必要があります。たとえば、'10, 20, 5'のようになります。設定値は、 |
|
|
次の文字列のリスト:
|
非表示層の活性化関数を定義します。たとえば、'''NNET_ACTIVATIONS_BIPOLAR_SIG'', ''NNET_ACTIVATIONS_TANH'''のようにします。
層ごとに異なる活性化関数を用意できます。 デフォルト値は 活性化関数の数は、 ノート: すべての引用符は一重引用符です。2つの一重引用符は、SQL文の一重引用符エスケープするために使用します。 |
|
|
|
この設定により、重みがランダムに初期化されるリージョンの下限が指定されます。
|
|
|
|
この設定により、重みが初期化されるリージョンの上限が指定されます。 デフォルト値は |
|
|
正の整数 |
この設定により、ニューラル・ネットワーク・アルゴリズムにおける最大反復数が指定されます。 デフォルト値は |
|
|
|
ニューラル・ネットワーク・アルゴリズムの収束許容値の設定を定義します。 デフォルト値は |
NNET_REGULARIZER |
|
ニューラル・ネットワーク・アルゴリズムの正則化設定です。トレーニング行の合計数が50000より大きい場合、デフォルトは トレーニング行の合計数が50000以下の場合、デフォルトは |
|
|
|
取り分けておいたメソッドの保持率を定義します。 デフォルト値は |
|
|
値は正整数である必要があります。 |
デフォルト値は |
NNET_REG_LAMBDA |
TO_CHAR(numeric_expr >=0) |
L2正則化パラメータ・ラムダを定義します。これは、NNET_REGULARIZER_HELDASIDEと一緒に設定することはできません。
デフォルト値は |
ore.neural関数は、ore.frameのデータに対して回帰用のフィードフォワードニューラル・ネットワーク・モデルを構築します。これは、ノード数を指定可能な複数の非表示層をサポートします。各層には、複数の活性化関数のいずれかを設定できます。
出力層は、単一の数値カテゴリ・ターゲットまたはバイナリのカテゴリ・ターゲットです。出力層には、任意の活性化関数を設定できます。これには、デフォルトで線形活性化関数があります。
ore.neuralの出力は、タイプore.neuralのオブジェクトになります。
ore.neural関数に対する引数の詳細は、help(ore.neural)を呼び出してください。
ore.neural関数を使用したモデル化は、センサー・データなどのノイズ・データおよび複合データに適しています。そのようなデータに発生する可能性のある問題は次のとおりです。
-
ピクセル値など、(数値)予測子が多い可能性がある
-
ターゲットが離散値、実数値またはそのような値のベクターである可能性がある
-
トレーニング・データに(ノイズに対して堅牢な)エラーが含まれている可能性がある
-
スコアリングが高速
-
モデルの透過性が不要なため、モデルの解釈が困難
ニューラル・ネットワーク・モデリングの一般的なステップは次のとおりです。
- アーキテクチャの指定
- データの準備
- モデルの構築
- 停止条件(反復、許容範囲内の検証セットでのエラー)の指定
- モデルの統計結果の表示
- モデルの改善
例4-5 ニューラル・ネットワーク・モデルの構築
この例では、デフォルト値(hidden sizeを1など)でニューラル・ネットワーク・モデルを構築します。この例では、longleyデータセットのサブセットをデータベース・メモリーのore.frameオブジェクトに、オブジェクトtrainDataとしてプッシュします。次に、longleyの別のサブセットをオブジェクトtestDataとしてデータベースにプッシュします。この例では、trainDataを使用してモデルを構築し、testDataを使用して結果を予測します。
trainData <- ore.push(longley[1:11, ])
testData <- ore.push(longley[12:16, ])
fit <- ore.neural('Employed ~ GNP + Population + Year', data = trainData)
ans <- predict(fit, newdata = testData)
ansこの例のリスト
R> trainData <- ore.push(longley[1:11, ])
R> testData <- ore.push(longley[12:16, ])
R> fit <- ore.neural('Employed ~ GNP + Population + Year', data = trainData)
R> ans <- predict(fit, newdata = testData)
R> ans
pred_Employed
1 67.97452
2 69.50893
3 70.28098
4 70.86127
5 72.31066
Warning message:
ORE object has no unique key - using random order
例4-6 ore.neuralの使用方法および活性化の指定
この例では、irisデータセットを一時データベース表にプッシュしますが、これにはプロキシore.frameオブジェクトのIRISが含まれています。この例では、ore.neural関数を使用してニューラル・ネットワーク・モデルを構築し、層ごとに異なる活性化関数を指定します。
IRIS <- ore.push(iris)
fit <- ore.neural(Petal.Length ~ Petal.Width + Sepal.Length,
data = IRIS,
hiddenSizes = c(20, 5),
activations = c("bSigmoid", "tanh", "linear"))
ans <- predict(fit, newdata = IRIS,
supplemental.cols = c("Petal.Length"))
options(ore.warn.order = FALSE)
head(ans, 3)
summary(ans)この例のリスト
R> IRIS <- ore.push(iris)
R> fit <- ore.neural(Petal.Length ~ Petal.Width + Sepal.Length,
+ data = IRIS,
+ hiddenSizes = c(20, 5),
+ activations = c("bSigmoid", "tanh", "linear"))
R>
R> ans <- predict(fit, newdata = IRIS,
+ supplemental.cols = c("Petal.Length"))
R> options(ore.warn.order = FALSE)
R> head(ans, 3)
Petal.Length pred_Petal.Length
1 1.4 1.416466
2 1.4 1.363385
3 1.3 1.310709
R> summary(ans)
Petal.Length pred_Petal.Length
Min. :1.000 Min. :1.080
1st Qu.:1.600 1st Qu.:1.568
Median :4.350 Median :4.346
Mean :3.758 Mean :3.742
3rd Qu.:5.100 3rd Qu.:5.224
Max. :6.900 Max. :6.3004.1.6 ランダム・フォレスト・モデルの構築
ore.randomForest関数は、ore.frameオブジェクト内のデータを分類するためのアンサンブル学習手法を提供します。
関数ore.randomForestは、データベース・サーバーで並行してツリーを成長させることにより、ランダム・フォレスト・モデルを構築します。これは、多数のデシジョン・ツリーを構築し、個々のツリーのクラスのモードであるクラスを出力します。この関数では、デシジョン・ツリーの一般的な問題であるオーバーフィットが回避されます。
Leo BreimanとAdele Cutlerによって開発されたランダム・フォレスト・アルゴリズムでは、バギングの概念と変数のランダムな選択を組み合せることにより、分散が制御されたデシジョン・ツリーの集合が生成されます。ランダム・フォレスト・アルゴリズムは高い精度を備えていますが、大規模なデータセットについては、パフォーマンスとスケーラビリティが問題になることがあります。
関数ore.randomForestは、モデルの構築およびスコアリングのために並行して実行されます。パラレル実行は、Oracle R Distribution (ORD)のrandomForestパッケージを使用している場合にも、オープン・ソースのrandomForestパッケージ4.6-10を使用している場合にも、発生することがあります。ore.randomForestおよびORDを使用すると、オープン・ソースのパッケージとともにore.randomForestを使用するよりも、必要なメモリーが少なくなる可能性があります。オープン・ソースのrandomForestパッケージを使用すると、Oracle Machine Learning for Rでは警告が発行されます。
関数ore.randomForestは、グローバル・オプションore.parallelを使用して、採用する並列度を決定します。この関数は、ore.randomForestオブジェクトを返します。
ore.randomForestオブジェクトに対するスコアリング・メソッドpredictの呼出しも、データベース・サーバーで並行して実行されます。cache.model引数では、予測中にランダム・フォレスト・モデル全体をメモリーにキャッシュするかどうかを指定します。使用可能なメモリーが十分ある場合は、パフォーマンスを向上させるために、cache.modelのデフォルト値であるTRUEを使用してください。
grabTreeメソッドは、指定されたツリーに関する情報を格納するore.frameオブジェクトを返します。ore.frameの各行は、ツリーの1つのノードを表します。
ノート:
関数ore.randomForestは、並行して実行されている各埋込みRセッションのトレーニング・データのコピーをロードします。大規模なデータセットの場合、これは、使用可能なメモリーの量を超えることがあります。パラレル・プロセスの数および使用可能なメモリーの量を適宜調整することをお薦めします。グローバル・オプションore.parallelで、パラレル・プロセスの数を指定します。埋込みRの実行プロセスで使用されるメモリーの量を制御する方法の詳細は、『Oracle Machine Learning for Rインストレーションおよび管理ガイド』の埋込みRが使用するメモリーの制御に関する項を参照してください。
ランダム・フォレスト・モデルの設定
次の表に、ランダム・フォレスト・モデルに適用される設定を示します。
表4-4 ランダム・フォレスト・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
ノードでの分割を選択する際に考慮する列のランダムなサブセットのサイズ。各ノードで、プールのサイズは同じですが、特定の候補列が変更されます。デフォルトは、モデル・シグネチャの列の半分です。特殊な値である |
|
|
|
フォレスト内のツリーの数 デフォルトは |
|
|
|
個々のツリーの作成で使用するためにランダムにサンプリングされるトレーニング・データの割合。デフォルトは、トレーニング・データ内の行数の半分です。 |
例4-7 ore.randomForestの使用方法
# Using the iris dataset
IRIS <- ore.push(iris)
mod <- ore.randomForest(Species~., IRIS)
tree10 <- grabTree(mod, k = 10, labelVar = TRUE)
ans <- predict(mod, IRIS, type="all", supplemental.cols="Species")
table(ans$Species, ans$prediction)
# Using the infert dataset
INFERT <- ore.push(infert)
formula <- case ~ age + parity + education + spontaneous + induced
rfMod <- ore.randomForest(formula, INFERT, ntree=1000, nodesize = 2)
tree <- grabTree(rfMod, k = 500)
rfPred <- predict(rfMod, INFERT, supplemental.cols = "case")
confusion.matrix <- with(rfPred, table(case, prediction))
confusion.matrixこの例のリスト
R> # Using the iris dataset
R> IRIS <- ore.push(iris)
R> mod <- ore.randomForest(Species~., IRIS)
R> tree10 <- grabTree(mod, k = 10, labelVar = TRUE)
R> ans <- predict(mod, IRIS, type="all", supplemental.cols="Species")
R> table(ans$Species, ans$prediction)
setosa versicolor virginica
setosa 50 0 0
versicolor 0 50 0
virginica 0 0 50
# Using the infert dataset
R> INFERT <- ore.push(infert)
R> formula <- case ~ age + parity + education + spontaneous + induced
R>
R> rfMod <- ore.randomForest(formula, INFERT, ntree=1000, nodesize = 2)
R> tree <- grabTree(rfMod, k = 500)
R>
R> rfPred <- predict(rfMod, INFERT, supplemental.cols = "case")
R>
R> confusion.matrix <- with(rfPred, table(case, prediction))
R> confusion.matrix
prediction
case 0 1
0 154 11
1 27 564.2 Oracle Machine Learning for SQLのモデルの構築
Oracle Machine Learning for RのOREdmパッケージの関数を使用して、RでOracle Machine Learning for SQLのモデルを構築します。
これらの関数については、次の各トピックで説明します。
- OML4Rを使用したOML4SQLモデルの構築について
Oracle Machine Learning for SQLの関数では、表、ビュー、スター・スキーマ、トランザクション・データおよび非構造化データを処理できます。 - 相関ルール
ore.odmAssocRules関数は、Aprioriアルゴリズムを実装することで、高頻度アイテム・セットを検索して相関モデルを生成します。 - 属性評価モデルの構築
属性評価では、ターゲットの予測における重要度に従って属性がランク付けされます。 - デシジョン・ツリー
ore.odmDT関数は、条件付き確率に基づいたOML4SQLのデシジョン・ツリー・アルゴリズムを使用します。 - 期待値最大化
ore.odmEM関数は、OML4SQLの期待値最大化(EM)アルゴリズムを使用するモデルを作成します。 - 明示的セマンティック分析
ore.odmESA関数は、OML4SQLの明示的セマンティック分析(ESA)アルゴリズムを使用するモデルを作成します。 - 拡張可能Rアルゴリズム・モデルの構築
ore.odmRAlg関数は、OML4SQLを使用して拡張可能Rアルゴリズム・モデルを作成します。 - 一般化線形モデル
ore.odmGLM関数は一般化線形モデル(GLM)を構築しますが、これは線形モデル(線形回帰)のクラスを含み、このクラスを拡張したものです。 - k-Means
ore.odmKM関数はOML4SQLのk-Means (KM)アルゴリズムを使用します。このアルゴリズムは、指定した数のクラスタにデータをパーティション化する、距離ベースのクラスタリング・アルゴリズムです。 - Naive Bayes
ore.odmNB関数は、OML4SQLのNaive Bayesモデルを構築します。 - Non-Negative Matrix Factorization
ore.odmNMF関数は、特徴抽出用にOML4SQLのNon-Negative Matrix Factorization (NMF)モデルを構築します。 - 直交パーティショニング・クラスタ
ore.odmOC関数は、直交パーティショニング・クラスタ(O-Cluster)アルゴリズムを使用してOML4SQLモデルを構築します。 - 特異値分解
ore.odmSVD関数は、OML4SQLの特異値分解(SVD)アルゴリズムを使用するモデルを作成します。 - サポート・ベクター・マシン
ore.odmSVM関数は、OML4Rのサポート・ベクター・マシン(SVM)モデルを構築します。 - パーティション化されたモデルの構築
パーティション化されたモデルは、複数のサブモデル(データの各パーティションに1つ)で構成されるアンサンブル・モデルです。 - テキスト処理モデルの構築
テキスト処理モデルでは、ctx.settings引数を使用してOracle Text属性の設定を指定します。
4.2.1 OML4Rを使用したOML4SQLモデルの構築について
Oracle Machine Learning for SQLの関数では、表、ビュー、スター・スキーマ、トランザクション・データおよび非構造化データを処理できます。
これらのOREdmパッケージ関数は、対応する予測分析関数およびOML4SQL関数に、一般的なRの使用方法に適合した引数を使用するRインタフェースを提供します。
この項の内容は次のとおりです。
- OML4RでサポートされるOML4SQLモデル
- OML4R関数を使用したインデータベース・モデル名および名前の変更について
各OREdmのRモデル・オブジェクトで、スロットnameは、OREdm関数で生成された基礎となるOML4SQLモデルの名前です。 - モデル設定の指定
OREdmパッケージの関数にはOracle Machine Learning for SQLモデルの設定を指定する引数が含まれ、一部にはテキスト処理パラメータを設定する引数が含まれます。
4.2.1.1 OML4RでサポートされるOML4SQLモデル
OREdmパッケージの関数は、Oracle DatabaseのOracle Machine Learning for SQLのインデータベース機械学習機能へのアクセスを提供します。これらの関数を使用して、データベースにOML4SQLモデルを構築します。
次の表に、OML4SQLモデルを構築するOML4Rの関数および対応するOML4SQLのアルゴリズムと関数を示します。
表4-5 Oracle Machine Learning for Rモデルの関数
| OML4Rの関数 | OML4SQLのアルゴリズム | OML4SQLの関数 |
|---|---|---|
|
|
最小記述長 |
分類または回帰の属性評価 |
|
|
Apriori |
相関ルール |
|
|
デシジョン・ツリー |
分類 |
|
(12.2の機能) |
期待値最大化 |
クラスタリング |
|
(12.2の機能) |
明示的セマンティック分析 |
特徴抽出 |
|
|
一般化線形モデル |
分類および回帰 |
|
|
k-Means |
クラスタリング |
|
|
Naive Bayes |
分類 |
|
|
Non-Negative Matrix Factorization |
特徴抽出 |
|
|
直交パーティショニング・クラスタ(O-Cluster) |
クラスタリング |
|
(12.2の機能) |
拡張可能Rアルゴリズム |
相関ルール、属性評価、分類、クラスタリング、特徴抽出および回帰 |
|
(12.2の機能) |
特異値分解 |
特徴抽出 |
|
|
サポート・ベクター・マシン |
分類および回帰 |
親トピック: OML4Rを使用したOML4SQLモデルの構築について
4.2.1.2 OML4R関数を使用したインデータベース・モデル名および名前の変更について
各OREdmのRモデル・オブジェクトで、スロットnameは、OREdm関数で生成された基礎となるOML4SQLモデルの名前です。
デフォルトでは、OREdm関数を使用して構築されたモデルは一時オブジェクトであるため、OML4Rデータストアに明示的に保存されないか、作成時に明示的な名前で指定されないかぎり、Rセッション終了後は保持されません。一方、Data MinerまたはSQLを使用して構築されたOML4SQLモデルは、明示的に削除されるまで存在します。
Rプロキシ・オブジェクトを保存または保持できます。OREdm関数で生成されたモデル・オブジェクトを保存すると、このオブジェクトはRセッション全体で存在でき、対応するインデータベース機械学習モデル・オブジェクトを所定の場所に保持できます。OREdmモデルが存在する間、インデータベース・モデル・オブジェクトをエクスポートおよびインポートし、OML4Rオブジェクトとは無関係に使用できます。
odm.settingsのMODEL_NAMEパラメータを使用して、データベースで作成されたインデータベース・モデル・オブジェクトに明示的に名前を付けることができます。名前が付けられたインデータベース・モデル・オブジェクトは、Oracle Data MinerまたはSQLを使用して作成されたインデータベース・モデル・オブジェクトのようにデータベースに保持されます。
例4-8 MODEL_NAMEパラメータを使用したインデータベース・モデルのプロキシ・オブジェクトの明示的な名前付け
この例では、ランダム・フォレスト・モデルを構築し、明示的な設定を使用してモデルに名前を付ける方法を示します。この例では、odm.settingsのMODEL_NAMEパラメータを使用して、データベースで作成されたインデータベース・モデル・オブジェクトに明示的に名前を付けます。
ore.exec("BEGIN DBMS_DATA_MINING.DROP_MODEL(model_name=> 'RF_CLASSIFICATION_MODEL'); EXCEPTION WHEN others THEN null; END;")
settings = list(RFOR_MTRY = 3,
RFOR_NUM_TREES = 100,
RFOR_SAMPLING_RATIO =0.5,
model_name="RF_CLASSIFICATION_MODEL")
MOD2 <- ore.odmRF(AFFINITY_CARD~., DEMO_DF.train, odm.settings= settings)
RES2 <- predict(MOD2, DEMO_DF.test, type= c("class","raw"), norm.votes=TRUE, cache.model=TRUE,
supplemental.cols=c("CUST_ID", "AFFINITY_CARD", "BOOKKEEPING_APPLICATION", "BULK_PACK_DISKETTES",
"EDUCATION", "FLAT_PANEL_MONITOR", "HOME_THEATER_PACKAGE", "HOUSEHOLD_SIZE",
"OCCUPATION", "OS_DOC_SET_KANJI", "PRINTER_SUPPLIES", "YRS_RESIDENCE", "Y_BOX_GAMES"))前述のコードでは、RF_CLASSIFICATION_MODELという名前のモデルがすでに存在する場合は削除する必要があります。同じ名前のモデルを作成しようとすると例外がスローされるためです。RF_CLASSIFCIATION_MODELという名前のインデータベース・ランダム・フォレスト分類モデルは、指定された設定を使用して構築され、モデルは変数MOD2によって参照されます。予測は、ランダム・フォレスト・モデルMOD2とAFFINITY_CARDをテスト・データ・セットDEMO DF testの予測変数として使用して実行され、結果は変数RES2のローカル・セッションに格納され、モデルはデータベースに保持されます。
Rモデルが存在するか、モデルにユーザー指定の名前を割り当てた場合は、OML4SQLモデル名を使用して、次のような他のインタフェースを介してOML4SQLモデルにアクセスします:
-
SQLインタフェース(SQL*Plus、SQL Developerなど)
-
Oracle Data Miner
具体的には、このモデルはOML4SQLの予測機能とともに使用できます。
Oracle Data Minerを使用して、次のことを実行できます。
-
利用可能なモデルのリストを表示する
-
モデル・ビューを使用してモデルの詳細を調べる
-
変換されたデータを適切にスコアリングする
ノート:
R空間での明示的なユーザー実行の変換は、SQLスコアリングまたはOracle Data Minerには継承されません。さらに、SQLを使用して、モデルの詳細を調べるため、または変換されたデータを適切にスコアリングするためのモデルのリストも取得できます。親トピック: OML4Rを使用したOML4SQLモデルの構築について
4.2.1.3 モデル設定の指定
OREdmパッケージの関数にはOracle Machine Learning for SQLモデルの設定を指定する引数が含まれ、一部にはテキスト処理パラメータを設定する引数が含まれます。
一般的なパラメータ設定
OREdm関数のodm.settings引数を使用すると、OML4SQLパラメータ設定のリストを指定できます。各リストの要素の名前および値は、それぞれパラメータ設定名および値を示します。設定値は数値または文字列である必要があります。各アルゴリズムの有効な設定については、『Oracle Machine Learning for SQLユーザーズ・ガイド』のモデル設定の指定に関する項を参照してください。
settings関数は、モデルを構築するために使用される各OML4SQLパラメータ設定名と値のペアをリストするdata.frameを返します。
テキスト処理属性設定
一部のOREdm関数には、Oracle Text属性固有の設定を指定できるテキスト処理属性設定を指定するctx.settings引数が含まれます。odm.settings引数を使用する場合、Oracleテキスト・ポリシー、各トークンが発生するドキュメントの最小数およびテキスト処理用の個別の特徴の最大数を指定できます。ctx.settings引数を使用する場合、テキストとして処理する列および適用するテキスト変換のタイプを指定します。
ctx.settings引数は、次の関数に適用されます。
-
ore.odmESA, 明示的セマンティック分析 -
ore.odmGLM, 一般化線形モデル -
ore.odmKMeans, k-Means -
ore.odmNMF, Non-Negative Matrix Factorization -
ore.odmSVD, 特異値分解 -
ore.odmSVM, サポート・ベクター・マシン
ノート:
Oracle Textポリシーを作成するには、ユーザーにはCTXSYS.CTX_DDL権限が必要です。
関連項目:
有効なテキスト属性値については、『Oracle Machine Learning for SQLユーザーズ・ガイド』のテキストに対する機械学習操作を含むモデルの作成に関する項を参照してください。親トピック: OML4Rを使用したOML4SQLモデルの構築について
4.2.2 相関ルール
ore.odmAssocRules関数は、Aprioriアルゴリズムを実装することで、高頻度アイテム・セットを検索して相関モデルを生成します。
この関数は、マーケット・バスケット分析などの大量のトランザクション・データにおいて、アイテムの同時発生を検出します。相関ルールは、トランザクション・レコードで一連のアイテムが出現すれば、もう1つの一連のアイテムが存在するというデータのパターンを特定します。ルールの形成に使用されるアイテムのグループは、発生(ルールの支持度)する頻度および後件が前件(ルールの信頼度)に従う回数に応じて、最小しきい値を渡す必要があります。相関モデルは、ユーザー指定のしきい値よりも大きい支持度および信頼度を備えるすべてのルールを生成します。Aprioriアルゴリズムは効率的で、トランザクション数、アイテム数および生成されるアイテム・セットおよびルールの数が有効に測定されます。
formulaの指定には~ termsという形式があり、ここでtermsは分析に含まれる一連の列名です。複数の列名は列名の間に+を使用することで指定されます。~ .は、dataのすべての列をモデル構築に使用する場合に使用します。列を除外するには、除外する各列名の前に-を使用します。関数は変換を確認するためにtermsの項目に適用できます。
ore.odmAssocRules関数は、次の形式のデータを受け入れます。
-
トランザクション・データ
-
アイテムIDおよびアイテム値を使用した複数レコードのケース・データ
-
リレーショナル・データ
データの形式の指定の例および関数の引数の詳細は、help(ore.odmAssocRules)をコールしてください。
関数rulesは、クラスore.rules (一連の相関ルールを指定します)のオブジェクトを返します。ore.pullを使用して、ore.rulesオブジェクトをローカルのRセッションのメモリーにプルできます。ローカルのインメモリー・オブジェクトは、arulesパッケージで定義されているクラスrulesのものです。help(ore.rules)を参照してください。
関数itemsetsは、クラスore.itemsets (一連のアイテムセットを指定します)のオブジェクトを返します。ore.pullを使用して、ore.itemsetsオブジェクトをローカルのRセッションのメモリーにプルできます。ローカルのインメモリー・オブジェクトは、arulesパッケージで定義されているクラスitemsetsのものです。help(ore.itemsets)を参照してください。
相関ルール・モデルの設定
次の表に、相関ルール・モデルに適用される設定を示します。
表4-6 相関ルール・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
ASSO_ABS_ERROR |
|
相関ルールのサンプリングの絶対誤差を指定します。
デフォルト値は |
|
|
|
集計する列を指定します。これは、集計する列の名前を含む文字列のカンマ区切りリストです。リスト内の列の数は10以下にする必要があります。
項目値は必須ではありません。 デフォルト値は 項目ごとに、集計する複数の列を指定できます。ただし、これを行うと、追加のデータをバッファするためにより多くのメモリーが必要になるとともに、入力データセットが大きくなり、操作が増えることからパフォーマンスにも影響が生じます。 |
|
|
|
前件の包含ルールを設定します。これは、文字列のカンマ区切りリストです。そのうちの少なくとも1つの文字列が、レポートされる各相関ルールの前件部分に出現する必要があります。 デフォルト値は |
|
|
|
前件の除外ルールを設定します。これは、文字列のカンマ区切りリストです。そのうちのいずれの文字列も、レポートされる各相関ルールの前件部分に出現できません。 デフォルト値は |
|
|
|
相関ルールのサンプルに対する信頼水準を指定します。
|
|
|
|
後件の包含ルールを設定します。これは、文字列のカンマ区切りリストです。そのうちの少なくとも1つの文字列が、レポートされる各相関ルールの後件部分に出現する必要があります。 デフォルト値は |
|
|
|
後件の除外ルールを設定します。これは、文字列のカンマ区切りリストです。そのうちのいずれの文字列も、レポートされる相関ルールの後件部分に出現できません。 除外ルールを使用すると、格納する必要があるデータを削減できますが、異なる包含ルールまたは除外ルールを実行するための追加のモデルを構築する必要が生じることがあります。 デフォルト値は |
ASSO_EX_RULES |
|
各相関ルールに適用される除外ルールを設定します。これは、相関ルールに出現できない文字列のカンマ区切りリストです。リスト内の項目をルールに含めることはできません。 デフォルト値は |
|
|
|
各相関ルールに適用される包含ルールを設定します。これは、文字列のカンマ区切りリストです。そのうちの少なくとも1つの文字列が、レポートされる各相関ルールに前件または後件として出現する必要があります デフォルト値は |
ASSO_MAX_RULE_LENGTH |
TO_CHAR( 2<= numeric_expr <=20) |
相関ルールの最大ルール長。 デフォルト値は |
ASSO_MIN_CONFIDENCE |
TO_CHAR( 0<= numeric_expr <=1) |
相関ルールの最小信頼度。 デフォルト値は |
ASSO_MIN_REV_CONFIDENCE |
TO_CHAR( 0<= numeric_expr <=1) |
各ルールで満たす必要がある最小逆確信度を設定します。 ルールの逆確信度は、ルールが発生するトランザクションの数を後件が発生するトランザクションの数で割ったものとして定義されています。 値は0から1の実数になります。 デフォルト値は |
|
|
TO_CHAR( 0<= numeric_expr <=1) |
相関ルールの最小支持度。 デフォルト値は |
|
|
TO_CHAR( 0<= numeric_expr <=1) |
各ルールで満たす必要がある絶対最小支持度。値は整数である必要があります。 デフォルト値は |
|
|
||
|
|
column_name |
トランザクションの項目を含む列の名前。この設定を指定すると、アルゴリズムは、次の2つの列で構成されるネイティブ・トランザクション形式でデータが表されると想定します。
|
ODMS_ITEM_VALUE_COLUMN_ NAME |
column_name |
トランザクションの各項目に関連付けられている値を含む列の名前。この設定は、データがネイティブ・トランザクション形式で表されることを示す
この項目値列では、(リンゴ3個などの)項目数、(マッキントッシュのリンゴなどの)項目タイプなどの情報を指定できます。 |
例4-9 ore.odmAssocRules関数の使用方法
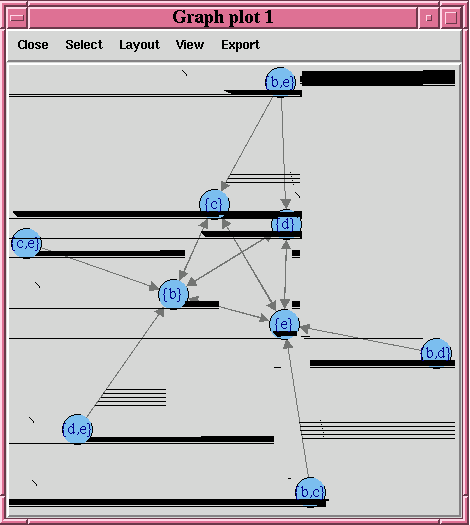
この例では、トランザクション・データセットに基づいて相関モデルを構築します。パッケージarulesおよびarulesVizは、生成されるルールおよびアイテムセットをクライアントのRセッションのメモリーにプルして表示するために必要です。ルールのグラフは、例の後の図に示されています。
# Load the arules and arulesViz packages.
library(arules)
library(arulesViz)
# Create some transactional data.
id <- c(1, 1, 1, 2, 2, 2, 2, 3, 3, 3, 3)
item <- c("b", "d", "e", "a", "b", "c", "e", "b", "c", "d", "e")
# Push the data to the database as an ore.frame object.
transdata_of <- ore.push(data.frame(ID = id, ITEM = item))
# Build a model with specifications.
ar.mod1 <- ore.odmAssocRules(~., transdata_of, case.id.column = "ID",
item.id.column = "ITEM", min.support = 0.6, min.confidence = 0.6,
max.rule.length = 3)
# Generate itemsets and rules of the model.
itemsets <- itemsets(ar.mod1)
rules <- rules(ar.mod1)
# Convert the rules to the rules object in arules package.
rules.arules <- ore.pull(rules)
inspect(rules.arules)
# Convert itemsets to the itemsets object in arules package.
itemsets.arules <- ore.pull(itemsets)
inspect(itemsets.arules)
# Plot the rules graph.
plot(rules.arules, method = "graph", interactive = TRUE)この例のリスト
R> # Load the arules and arulesViz packages.
R> library(arules)
R> library(arulesViz)
R> # Create some transactional data.
R> id <- c(1, 1, 1, 2, 2, 2, 2, 3, 3, 3, 3)
R> item <- c("b", "d", "e", "a", "b", "c", "e", "b", "c", "d", "e")
R> # Push the data to the database as an ore.frame object.
R> transdata_of <- ore.push(data.frame(ID = id, ITEM = item))
R> # Build a model with specifications.
R> ar.mod1 <- ore.odmAssocRules(~., transdata_of, case.id.column = "ID",
+ item.id.column = "ITEM", min.support = 0.6, min.confidence = 0.6,
+ max.rule.length = 3)
R> # Generate itemsets and rules of the model.
R> itemsets <- itemsets(ar.mod1)
R> rules <- rules(ar.mod1)
R> # Convert the rules to the rules object in arules package.
R> rules.arules <- ore.pull(rules)
R> inspect(rules.arules)
lhs rhs support confidence lift
1 {b} => {e} 1.0000000 1.0000000 1
2 {e} => {b} 1.0000000 1.0000000 1
3 {c} => {e} 0.6666667 1.0000000 1
4 {d,
e} => {b} 0.6666667 1.0000000 1
5 {c,
e} => {b} 0.6666667 1.0000000 1
6 {b,
d} => {e} 0.6666667 1.0000000 1
7 {b,
c} => {e} 0.6666667 1.0000000 1
8 {d} => {b} 0.6666667 1.0000000 1
9 {d} => {e} 0.6666667 1.0000000 1
10 {c} => {b} 0.6666667 1.0000000 1
11 {b} => {d} 0.6666667 0.6666667 1
12 {b} => {c} 0.6666667 0.6666667 1
13 {e} => {d} 0.6666667 0.6666667 1
14 {e} => {c} 0.6666667 0.6666667 1
15 {b,
e} => {d} 0.6666667 0.6666667 1
16 {b,
e} => {c} 0.6666667 0.6666667 1
R> # Convert itemsets to the itemsets object in arules package.
R> itemsets.arules <- ore.pull(itemsets)
R> inspect(itemsets.arules)
items support
1 {b} 1.0000000
2 {e} 1.0000000
3 {b,
e} 1.0000000
4 {c} 0.6666667
5 {d} 0.6666667
6 {b,
c} 0.6666667
7 {b,
d} 0.6666667
8 {c,
e} 0.6666667
9 {d,
e} 0.6666667
10 {b,
c,
e} 0.6666667
11 {b,
d,
e} 0.6666667
R> # Plot the rules graph.
R> plot(rules.arules, method = "graph", interactive = TRUE)
4.2.3 属性評価モデルの構築
属性評価では、ターゲットの予測における重要度に従って属性がランク付けされます。
ore.odmAI関数は、OML4SQLの最小記述長アルゴリズムを使用して属性評価を計算します。最小記述長(MDL)は、情報理論モデルの選択原理の1つである。それは、情報理論(情報の定量化の研究)および学習理論(経験的データに基づく一般化の容量の研究)における重要な概念です。
MDLでは、最も単純でコンパクトな表現が、データの説明として最適かつ最も可能性が高いとみなされます。MDL原理は、OML4SQLの属性評価モデルの作成に使用されます。
OML4SQLを使用して構築された属性評価モデルは、新しいデータに適用できません。
ore.odmAI関数は、属性のランキングおよび属性評価の値を作成します。
ノート:
OREdm属性評価モデルは、モデル・オブジェクトが保持されない、およびRモデル・オブジェクトが返されないという点で、OML4SQL属性評価モデルとは異なります。モデルによって作成された重要度ランキングのみが返されます。
ore.odmAI関数の引数の詳細は、help(ore.odmAI)を呼び出してください。
例4-10 ore.odmAI関数の使用方法
この例では、data.frameのirisをore.frameのiris_ofとしてデータベースにプッシュします。この例では次に、属性評価モデルを構築します。
iris_of <- ore.push(iris) ore.odmAI(Species ~ ., iris_of)
この例のリスト
R> iris_of <- ore.push(iris)
R> ore.odmAI(Species ~ ., iris_of)
Call:
ore.odmAI(formula = Species ~ ., data = iris_of)
Importance:
importance rank
Petal.Width 1.1701851 1
Petal.Length 1.1494402 2
Sepal.Length 0.5248815 3
Sepal.Width 0.2504077 44.2.4 デシジョン・ツリー
ore.odmDT関数は、条件付き確率に基づいたOML4SQLのデシジョン・ツリー・アルゴリズムを使用します。
デシジョン・ツリー・モデルは分類モデルです。デシジョン・ツリーはルールを生成します。ルールは、ユーザーが容易に理解でき、レコード・セットを識別するためにデータベース内で使用できる条件文です。
デシジョン・ツリーは、一連の質問を問うことによってターゲット値を予測します。各段階で問われる質問はそれぞれ、直前の質問に対する回答によって決まります。最終的に特定のターゲット値を一意に識別できるような質問を重ねていきます。図形的には、このプロセスがツリー構造を形成します。
デシジョン・ツリー・アルゴリズムは、トレーニング・プロセスにおいて、ケース(レコード)のセットを2つの子ノードに分割する最も効率的な方法を繰り返し見つける必要があります。ore.odmDT関数では、この分岐の計算用に2つの同種メトリック(giniおよびentropy)を使用できます。デフォルトのメトリックはジニです。
ore.odmDT関数の引数の詳細は、help(ore.odmDT)をコールしてください。
デシジョン・ツリー・モデルの設定
次の表は、デシジョン・ツリー・モデルに適用される設定のリストです。
表4-7 デシジョン・ツリー・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
デシジョン・ツリーのツリー不純メトリック。 ツリー・アルゴリズムでは、各ノードでのデータの分岐に最適なテスト質問が検索されます。最適な分岐および分岐値は、ノードのエンティティに対するターゲット値の同質性(純度)が最大限に高くなるものです。純度はメトリックによるものです。デシジョン・ツリーでは、純度メトリックとしてジニ( |
|
|
デシジョン・ツリーの場合:
ランダム・フォレストの場合:
|
分岐の条件: ツリーの最大深度(ルート・ノードとリーフ・ノードとの間(リーフ・ノードを含む)の最大ノード数)。 デシジョン・ツリーの場合、デフォルトは ランダム・フォレストの場合、デフォルトは |
|
|
|
トレーニング・データ内の行の割合として表現されたノード内のトレーニング行の最小数。
デフォルトは |
|
|
|
トレーニング行の割合として表現された、ノードの分割を検討するために必要な行の最小数。
デフォルトは |
|
|
|
ノード内の行の最小数。 デフォルトは |
|
|
|
分岐の条件: 値として表現される親ノードのレコードの最小数。レコード数がこの値よりも少ない場合、分岐は試行されません。 デフォルトは |
例4-11 ore.odmDT関数の使用方法
この例では、入力ore.frameを作成し、モデルを構築して、予測を行い、混同マトリクスを生成します。
m <- mtcars m$gear <- as.factor(m$gear) m$cyl <- as.factor(m$cyl) m$vs <- as.factor(m$vs) m$ID <- 1:nrow(m) mtcars_of <- ore.push(m) row.names(mtcars_of) <- mtcars_of # Build the model. dt.mod <- ore.odmDT(gear ~ ., mtcars_of) summary(dt.mod) # Make predictions and generate a confusion matrix. dt.res <- predict (dt.mod, mtcars_of, "gear") with(dt.res, table(gear, PREDICTION))
この例のリスト
R> m <- mtcars
R> m$gear <- as.factor(m$gear)
R> m$cyl <- as.factor(m$cyl)
R> m$vs <- as.factor(m$vs)
R> m$ID <- 1:nrow(m)
R> mtcars_of <- ore.push(m)
R> row.names(mtcars_of) <- mtcars_of
R> # Build the model.
R> dt.mod <- ore.odmDT(gear ~ ., mtcars_of)
R> summary(dt.mod)
Call:
ore.odmDT(formula = gear ~ ., data = mtcars_of)
n = 32
Nodes:
parent node.id row.count prediction split
1 NA 0 32 3 <NA>
2 0 1 16 4 (disp <= 196.299999999999995)
3 0 2 16 3 (disp > 196.299999999999995)
surrogate full.splits
1 <NA> <NA>
2 (cyl in ("4" "6" )) (disp <= 196.299999999999995)
3 (cyl in ("8" )) (disp > 196.299999999999995)
Settings:
value
prep.auto on
impurity.metric impurity.gini
term.max.depth 7
term.minpct.node 0.05
term.minpct.split 0.1
term.minrec.node 10
term.minrec.split 20
R> # Make predictions and generate a confusion matrix.
R> dt.res <- predict (dt.mod, mtcars_of, "gear")
R> with(dt.res, table(gear, PREDICTION))
PREDICTION
gear 3 4
3 14 1
4 0 12
5 2 34.2.5 期待値最大化
ore.odmEM関数は、OML4SQLの期待値最大化(EM)アルゴリズムを使用するモデルを作成します。
EMは、確率的クラスタリングを実行する密度推定アルゴリズムです。密度推定の目的は、所与の母集団がどのように分布しているかを取得する密度関数を構成することです。密度推定値は、母集団のサンプルを表現する観測済データに基づく。
ore.odmEM関数の引数の詳細は、help(ore.odmEM)をコールしてください。
期待値の最大化モデルの設定
次の表に、期待値最大化モデルに適用される設定を示します。
表4-8 期待値最大化モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
モデルに相関していない属性を含めるかどうかの指定。 ノート: この設定は、ネストしていない属性にのみ適用されます。デフォルトは、システムによって決定されます。 |
|
|
|
モデルに含める相関属性の最大数。 ノート: この設定は、ネストしていない(2D)属性にのみ適用されます。デフォルト値は |
|
|
|
量的属性をモデリングする分布。入力表または入力ビューの全体に適用され、属性ごとの指定は許可されません。
オプションには、ベルヌーイ分布、ガウス分布またはシステム決定の分布が含まれます。ベルヌーイ分布やガウス分布を選択した場合、すべての量的属性が同じタイプの分布を使用してモデリングされます。システム決定の分布の場合は、個々の属性で、データに応じて異なる分布(ベルヌーイ分布またはガウス分布)を使用できます。 デフォルト値は |
|
|
|
量的列のクラスタ統計を収集するために使用される等幅ビンの数。
デフォルトは |
|
|
|
ネストした各列で使用される予測の数を指定します。指定した数の予測よりも列の個別属性の数が少ない場合、データの予測は行われません。この設定は、すべてのネストしている列に適用されます。 デフォルトは |
|
|
|
複数値のベルヌーイ分布において、量的列のモデリングに使用されるクオンタイル・ビンの数を指定します。 デフォルトは、システムによって決定されます。 |
|
|
|
複数値のベルヌーイ分布において、質的列のモデリングに使用される上位Nビンの数を指定します。 デフォルトは、システムによって決定されます。 |
例4-12 ore.odmEM関数の使用方法
## Synthetic 2-dimensional data set
set.seed(7654)
x <- rbind(matrix(rnorm(100, mean = 4, sd = 0.3), ncol = 2),
matrix(rnorm(100, mean = 2, sd = 0.3), ncol = 2))
colnames(x) <- c("x", "y")
X <- ore.push (data.frame(ID=1:100,x))
rownames(X) <- X$ID
em.mod <- NULL
em.mod <- ore.odmEM(~., X, num.centers = 2L)
summary(em.mod)
rules(em.mod)
clusterhists(em.mod)
histogram(em.mod)
em.res <- predict(em.mod, X, type="class", supplemental.cols=c("x", "y"))
head(em.res)
em.res.local <- ore.pull(em.res)
plot(data.frame(x=em.res.local$x, y=em.res.local$y), col=em.res.local$CLUSTER_ID)
points(em.mod$centers2, col = rownames(em.mod$centers2), pch=8, cex=2)
head(predict(em.mod,X))
head(predict(em.mod,X,type=c("class","raw")))
head(predict(em.mod,X,type=c("class","raw"),supplemental.cols=c("x","y")))
head(predict(em.mod,X,type="raw",supplemental.cols=c("x","y")))この例のリスト
R> ## Synthetic 2-dimensional data set
R>
R> set.seed(7654)
R>
R> x <- rbind(matrix(rnorm(100, mean = 4, sd = 0.3), ncol = 2),
+ matrix(rnorm(100, mean = 2, sd = 0.3), ncol = 2))
R> colnames(x) <- c("x", "y")
R>
R> X <- ore.push (data.frame(ID=1:100,x))
R> rownames(X) <- X$ID
R>
R> em.mod <- NULL
R> em.mod <- ore.odmEM(~., X, num.centers = 2L)
R>
R> summary(em.mod)
Call:
ore.odmEM(formula = ~., data = X, num.centers = 2L)
Settings:
value
clus.num.clusters 2
cluster.components cluster.comp.enable
cluster.statistics clus.stats.enable
cluster.thresh 2
linkage.function linkage.single
loglike.improvement .001
max.num.attr.2d 50
min.pct.attr.support .1
model.search model.search.disable
num.components 20
num.distribution num.distr.system
num.equiwidth.bins 11
num.iterations 100
num.projections 50
random.seed 0
remove.components remove.comps.enable
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto ON
Centers:
MEAN.ID MEAN.x MEAN.y
2 25.5 4.03 3.96
3 75.5 1.93 1.99
R> rules(em.mod)
cluster.id rhs.support rhs.conf lhr.support lhs.conf lhs.var lhs.var.support lhs.var.conf predicate
1 1 100 1.0 100 1.00 ID 100 0.0000 ID <= 100
2 1 100 1.0 100 1.00 ID 100 0.0000 ID >= 1
3 1 100 1.0 100 1.00 x 100 0.2500 x <= 4.6298
4 1 100 1.0 100 1.00 x 100 0.2500 x >= 1.3987
5 1 100 1.0 100 1.00 y 100 0.3000 y <= 4.5846
6 1 100 1.0 100 1.00 y 100 0.3000 y >= 1.3546
7 2 50 0.5 50 1.00 ID 50 0.0937 ID <= 50.5
8 2 50 0.5 50 1.00 ID 50 0.0937 ID >= 1
9 2 50 0.5 50 1.00 x 50 0.0937 x <= 4.6298
10 2 50 0.5 50 1.00 x 50 0.0937 x > 3.3374
11 2 50 0.5 50 1.00 y 50 0.0937 y <= 4.5846
12 2 50 0.5 50 1.00 y 50 0.0937 y > 2.9696
13 3 50 0.5 50 0.98 ID 49 0.0937 ID <= 100
14 3 50 0.5 50 0.98 ID 49 0.0937 ID > 50.5
15 3 50 0.5 49 0.98 x 49 0.0937 x <= 2.368
16 3 50 0.5 49 0.98 x 49 0.0937 x >= 1.3987
17 3 50 0.5 49 0.98 y 49 0.0937 y <= 2.6466
18 3 50 0.5 49 0.98 y 49 0.0937 y >= 1.3546
R> clusterhists(em.mod)
cluster.id variable bin.id lower.bound upper.bound label count
1 1 ID 1 1.00 10.90 1:10.9 10
2 1 ID 2 10.90 20.80 10.9:20.8 10
3 1 ID 3 20.80 30.70 20.8:30.7 10
4 1 ID 4 30.70 40.60 30.7:40.6 10
5 1 ID 5 40.60 50.50 40.6:50.5 10
6 1 ID 6 50.50 60.40 50.5:60.4 10
7 1 ID 7 60.40 70.30 60.4:70.3 10
8 1 ID 8 70.30 80.20 70.3:80.2 10
9 1 ID 9 80.20 90.10 80.2:90.1 10
10 1 ID 10 90.10 100.00 90.1:100 10
11 1 ID 11 NA NA : 0
12 1 x 1 1.40 1.72 1.399:1.722 11
13 1 x 2 1.72 2.04 1.722:2.045 22
14 1 x 3 2.04 2.37 2.045:2.368 16
15 1 x 4 2.37 2.69 2.368:2.691 1
16 1 x 5 2.69 3.01 2.691:3.014 0
17 1 x 6 3.01 3.34 3.014:3.337 0
18 1 x 7 3.34 3.66 3.337:3.66 4
19 1 x 8 3.66 3.98 3.66:3.984 18
20 1 x 9 3.98 4.31 3.984:4.307 22
21 1 x 10 4.31 4.63 4.307:4.63 6
22 1 x 11 NA NA : 0
23 1 y 1 1.35 1.68 1.355:1.678 7
24 1 y 2 1.68 2.00 1.678:2.001 18
25 1 y 3 2.00 2.32 2.001:2.324 18
26 1 y 4 2.32 2.65 2.324:2.647 6
27 1 y 5 2.65 2.97 2.647:2.97 1
28 1 y 6 2.97 3.29 2.97:3.293 4
29 1 y 7 3.29 3.62 3.293:3.616 3
30 1 y 8 3.62 3.94 3.616:3.939 16
31 1 y 9 3.94 4.26 3.939:4.262 16
32 1 y 10 4.26 4.58 4.262:4.585 11
33 1 y 11 NA NA : 0
34 2 ID 1 1.00 10.90 1:10.9 10
35 2 ID 2 10.90 20.80 10.9:20.8 10
36 2 ID 3 20.80 30.70 20.8:30.7 10
37 2 ID 4 30.70 40.60 30.7:40.6 10
38 2 ID 5 40.60 50.50 40.6:50.5 10
39 2 ID 6 50.50 60.40 50.5:60.4 0
40 2 ID 7 60.40 70.30 60.4:70.3 0
41 2 ID 8 70.30 80.20 70.3:80.2 0
42 2 ID 9 80.20 90.10 80.2:90.1 0
43 2 ID 10 90.10 100.00 90.1:100 0
44 2 ID 11 NA NA : 0
45 2 x 1 1.40 1.72 1.399:1.722 0
46 2 x 2 1.72 2.04 1.722:2.045 0
47 2 x 3 2.04 2.37 2.045:2.368 0
48 2 x 4 2.37 2.69 2.368:2.691 0
49 2 x 5 2.69 3.01 2.691:3.014 0
50 2 x 6 3.01 3.34 3.014:3.337 0
51 2 x 7 3.34 3.66 3.337:3.66 4
52 2 x 8 3.66 3.98 3.66:3.984 18
53 2 x 9 3.98 4.31 3.984:4.307 22
54 2 x 10 4.31 4.63 4.307:4.63 6
55 2 x 11 NA NA : 0
56 2 y 1 1.35 1.68 1.355:1.678 0
57 2 y 2 1.68 2.00 1.678:2.001 0
58 2 y 3 2.00 2.32 2.001:2.324 0
59 2 y 4 2.32 2.65 2.324:2.647 0
60 2 y 5 2.65 2.97 2.647:2.97 0
61 2 y 6 2.97 3.29 2.97:3.293 4
62 2 y 7 3.29 3.62 3.293:3.616 3
63 2 y 8 3.62 3.94 3.616:3.939 16
64 2 y 9 3.94 4.26 3.939:4.262 16
65 2 y 10 4.26 4.58 4.262:4.585 11
66 2 y 11 NA NA : 0
67 3 ID 1 1.00 10.90 1:10.9 0
68 3 ID 2 10.90 20.80 10.9:20.8 0
69 3 ID 3 20.80 30.70 20.8:30.7 0
70 3 ID 4 30.70 40.60 30.7:40.6 0
71 3 ID 5 40.60 50.50 40.6:50.5 0
72 3 ID 6 50.50 60.40 50.5:60.4 10
73 3 ID 7 60.40 70.30 60.4:70.3 10
74 3 ID 8 70.30 80.20 70.3:80.2 10
75 3 ID 9 80.20 90.10 80.2:90.1 10
76 3 ID 10 90.10 100.00 90.1:100 10
77 3 ID 11 NA NA : 0
78 3 x 1 1.40 1.72 1.399:1.722 11
79 3 x 2 1.72 2.04 1.722:2.045 22
80 3 x 3 2.04 2.37 2.045:2.368 16
81 3 x 4 2.37 2.69 2.368:2.691 1
82 3 x 5 2.69 3.01 2.691:3.014 0
83 3 x 6 3.01 3.34 3.014:3.337 0
84 3 x 7 3.34 3.66 3.337:3.66 0
85 3 x 8 3.66 3.98 3.66:3.984 0
86 3 x 9 3.98 4.31 3.984:4.307 0
87 3 x 10 4.31 4.63 4.307:4.63 0
88 3 x 11 NA NA : 0
89 3 y 1 1.35 1.68 1.355:1.678 7
90 3 y 2 1.68 2.00 1.678:2.001 18
91 3 y 3 2.00 2.32 2.001:2.324 18
92 3 y 4 2.32 2.65 2.324:2.647 6
93 3 y 5 2.65 2.97 2.647:2.97 1
94 3 y 6 2.97 3.29 2.97:3.293 0
95 3 y 7 3.29 3.62 3.293:3.616 0
96 3 y 8 3.62 3.94 3.616:3.939 0
97 3 y 9 3.94 4.26 3.939:4.262 0
98 3 y 10 4.26 4.58 4.262:4.585 0
99 3 y 11 NA NA : 0
R> histogram(em.mod)
R>
R> em.res <- predict(em.mod, X, type="class", supplemental.cols=c("x", "y"))
R> head(em.res)
x y CLUSTER_ID
1 4.15 3.63 2
2 3.88 4.13 2
3 3.72 4.10 2
4 3.78 4.14 2
5 4.22 4.35 2
6 4.07 3.62 2
R> em.res.local <- ore.pull(em.res)
R> plot(data.frame(x=em.res.local$x, y=em.res.local$y), col=em.res.local$CLUSTER_ID)
R> points(em.mod$centers2, col = rownames(em.mod$centers2), pch=8, cex=2)
R>
R> head(predict(em.mod,X))
'2' '3' CLUSTER_ID
1 1 1.14e-54 2
2 1 1.63e-55 2
3 1 1.10e-51 2
4 1 1.53e-52 2
5 1 9.02e-62 2
6 1 3.20e-49 2
R> head(predict(em.mod,X,type=c("class","raw")))
'2' '3' CLUSTER_ID
1 1 1.14e-54 2
2 1 1.63e-55 2
3 1 1.10e-51 2
4 1 1.53e-52 2
5 1 9.02e-62 2
6 1 3.20e-49 2
R> head(predict(em.mod,X,type=c("class","raw"),supplemental.cols=c("x","y")))
'2' '3' x y CLUSTER_ID
1 1 1.14e-54 4.15 3.63 2
2 1 1.63e-55 3.88 4.13 2
3 1 1.10e-51 3.72 4.10 2
4 1 1.53e-52 3.78 4.14 2
5 1 9.02e-62 4.22 4.35 2
6 1 3.20e-49 4.07 3.62 2
R> head(predict(em.mod,X,type="raw",supplemental.cols=c("x","y")))
x y '2' '3'
1 4.15 3.63 1 1.14e-54
2 3.88 4.13 1 1.63e-55
3 3.72 4.10 1 1.10e-51
4 3.78 4.14 1 1.53e-52
5 4.22 4.35 1 9.02e-62
6 4.07 3.62 1 3.20e-494.2.6 明示的セマンティック分析
ore.odmESA関数は、OML4SQLの明示的セマンティック分析(ESA)アルゴリズムを使用するモデルを作成します。
ESAは、特徴抽出用にOML4SQLで使用される教師なしアルゴリズムです。ESAは、潜在的な特徴は検出しませんが、既存のナレッジ・ベースに基づく明示的特徴を使用します。
明示的ナレッジは、多くの場合テキスト・フォームで存在します。複数のナレッジ・ベースを、テキスト・ドキュメントのコレクションとして使用できます。これらのナレッジ・ベースは、Wikipediaなど汎用のものやドメイン固有のものの場合があります。データ準備は、テキストを、属性と概念の関連性を取得するベクターに変換します。
ore.odmESA関数の引数の詳細は、help(ore.odmESA)をコールしてください。
明示的セマンティック分析モデルの設定
次の表に、明示的セマンティック分析モデルに適用される設定を示します。
表4-9 明示的セマンティック分析モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
負でない数値 |
この設定により、変換される作成データの属性の重みに対して小さい値がしきい値処理されます。デフォルトは、 |
|
|
テキスト入力は テキスト以外の入力は |
この設定により、入力行に表示する必要があるゼロ以外のエントリの最小数が決まります。デフォルトはテキスト入力の場合は100、テキスト以外の入力の場合は0です。 |
|
|
正の整数 |
この設定により、属性ごとの特徴の最大数が制御されます。デフォルトは1000です。
|
例4-13 ore.odmESA関数の使用方法
title <- c('Aids in Africa: Planning for a long war',
'Mars rover maneuvers for rim shot',
'Mars express confirms presence of water at Mars south pole',
'NASA announces major Mars rover finding',
'Drug access, Asia threat in focus at AIDS summit',
'NASA Mars Odyssey THEMIS image: typical crater',
'Road blocks for Aids')
# TEXT contents in character column
df <- data.frame(CUST_ID = seq(length(title)), TITLE = title)
ESA_TEXT <- ore.push(df)
# TEXT contents in clob column
attr(df$TITLE, "ora.type") <- "clob"
ESA_TEXT_CLOB <- ore.push(df)
# Create text policy (CTXSYS.CTX_DDL privilege is required)
ore.exec("Begin ctx_ddl.create_policy('ESA_TXTPOL'); End;")
# Specify TEXT POLICY_NAME, MIN_DOCUMENTS, MAX_FEATURES and
# ESA algorithm settings in odm.settings
esa.mod <- ore.odmESA(~ TITLE, data = ESA_TEXT_CLOB,
odm.settings = list(case_id_column_name = "CUST_ID",
ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL",
ODMS_TEXT_MIN_DOCUMENTS = 1,
ODMS_TEXT_MAX_FEATURES = 3,
ESAS_MIN_ITEMS = 1,
ESAS_VALUE_THRESHOLD = 0.0001,
ESAS_TOPN_FEATURES = 3))
class(esa.mod)
summary(esa.mod)
settings(esa.mod)
features(esa.mod)
predict(esa.mod, ESA_TEXT, type = "class", supplemental.cols = "TITLE")
# Use ctx.settings to specify a character column as TEXT and
# the same settings as above as well as TOKEN_TYPE
esa.mod2 <- ore.odmESA(~ TITLE, data = ESA_TEXT,
odm.settings = list(case_id_column_name = "CUST_ID", ESAS_MIN_ITEMS = 1),
ctx.settings = list(TITLE =
"TEXT(POLICY_NAME:ESA_TXTPOL)(TOKEN_TYPE:STEM)(MIN_DOCUMENTS:1)(MAX_FEATURES:3)"))
summary(esa.mod2)
settings(esa.mod2)
features(esa.mod2)
predict(esa.mod2, ESA_TEXT_CLOB, type = "class", supplemental.cols = "TITLE")
ore.exec("Begin ctx_ddl.drop_policy('ESA_TXTPOL'); End;")この例のリスト
R> title <- c('Aids in Africa: Planning for a long war',
+ 'Mars rover maneuvers for rim shot',
+ 'Mars express confirms presence of water at Mars south pole',
+ 'NASA announces major Mars rover finding',
+ 'Drug access, Asia threat in focus at AIDS summit',
+ 'NASA Mars Odyssey THEMIS image: typical crater',
+ 'Road blocks for Aids')
R>
R> # TEXT contents in character column
R> df <- data.frame(CUST_ID = seq(length(title)), TITLE = title)
R> ESA_TEXT <- ore.push(df)
R>
R> # TEXT contents in clob column
R> attr(df$TITLE, "ora.type") <- "clob"
R> ESA_TEXT_CLOB <- ore.push(df)
R>
R> # Create a text policy (CTXSYS.CTX_DDL privilege is required)
R> ore.exec("Begin ctx_ddl.create_policy('ESA_TXTPOL'); End;")
R>
R> # Specify TEXT POLICY_NAME, MIN_DOCUMENTS, MAX_FEATURES and
R> # ESA algorithm settings in odm.settings
R> esa.mod <- ore.odmESA(~ TITLE, data = ESA_TEXT_CLOB,
+ odm.settings = list(case_id_column_name = "CUST_ID",
+ ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL",
+ ODMS_TEXT_MIN_DOCUMENTS = 1,
+ ODMS_TEXT_MAX_FEATURES = 3,
+ ESAS_MIN_ITEMS = 1,
+ ESAS_VALUE_THRESHOLD = 0.0001,
+ ESAS_TOPN_FEATURES = 3))
R> class(esa.mod)
[1] "ore.odmESA" "ore.model"
R> summary(esa.mod)
Call:
ore.odmESA(formula = ~TITLE, data = ESA_TEXT_CLOB, odm.settings = list(case_id_column_name = "CUST_ID",
ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL", ODMS_TEXT_MIN_DOCUMENTS = 1,
ODMS_TEXT_MAX_FEATURES = 3, ESAS_MIN_ITEMS = 1, ESAS_VALUE_THRESHOLD = 1e-04,
ESAS_TOPN_FEATURES = 3))
Settings:
value
min.items 1
topn.features 3
value.threshold 1e-04
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
odms.text.max.features 3
odms.text.min.documents 1
odms.text.policy.name ESA_TXTPOL
prep.auto ON
Features:
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 TITLE.AIDS <NA> 1.0000000
2 2 TITLE.MARS <NA> 0.4078615
3 2 TITLE.ROVER <NA> 0.9130438
4 3 TITLE.MARS <NA> 1.0000000
5 4 TITLE.NASA <NA> 0.6742695
6 4 TITLE.ROVER <NA> 0.6742695
7 5 TITLE.AIDS <NA> 1.0000000
8 6 TITLE.MARS <NA> 0.4078615
9 6 TITLE.NASA <NA> 0.9130438
10 7 TITLE.AIDS <NA> 1.0000000
R> settings(esa.mod)
SETTING_NAME SETTING_VALUE SETTING_TYPE
1 ALGO_NAME ALGO_EXPLICIT_SEMANTIC_ANALYS INPUT
2 ESAS_MIN_ITEMS 1 INPUT
3 ESAS_TOPN_FEATURES 3 INPUT
4 ESAS_VALUE_THRESHOLD 1e-04 INPUT
5 ODMS_MISSING_VALUE_TREATMENT ODMS_MISSING_VALUE_AUTO DEFAULT
6 ODMS_SAMPLING ODMS_SAMPLING_DISABLE DEFAULT
7 ODMS_TEXT_MAX_FEATURES 3 INPUT
8 ODMS_TEXT_MIN_DOCUMENTS 1 INPUT
9 ODMS_TEXT_POLICY_NAME ESA_TXTPOL INPUT
10 PREP_AUTO ON INPUT
R> features(esa.mod)
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 TITLE.AIDS <NA> 1.0000000
2 2 TITLE.MARS <NA> 0.4078615
3 2 TITLE.ROVER <NA> 0.9130438
4 3 TITLE.MARS <NA> 1.0000000
5 4 TITLE.NASA <NA> 0.6742695
6 4 TITLE.ROVER <NA> 0.6742695
7 5 TITLE.AIDS <NA> 1.0000000
8 6 TITLE.MARS <NA> 0.4078615
9 6 TITLE.NASA <NA> 0.9130438
10 7 TITLE.AIDS <NA> 1.0000000
R> predict(esa.mod, ESA_TEXT, type = "class", supplemental.cols = "TITLE")
TITLE FEATURE_ID
1 Aids in Africa: Planning for a long war 1
2 Mars rover maneuvers for rim shot 2
3 Mars express confirms presence of water at Mars south pole 3
4 NASA announces major Mars rover finding 4
5 Drug access, Asia threat in focus at AIDS summit 1
6 NASA Mars Odyssey THEMIS image: typical crater 6
7 Road blocks for Aids 1
R>
R> # Use ctx.settings to specify a character column as TEXT and
R> # the same settings as above as well as TOKEN_TYPE
R> esa.mod2 <- ore.odmESA(~ TITLE, data = ESA_TEXT,
+ odm.settings = list(case_id_column_name = "CUST_ID", ESAS_MIN_ITEMS = 1),
+ ctx.settings = list(TITLE =
+ "TEXT(POLICY_NAME:ESA_TXTPOL)(TOKEN_TYPE:STEM)(MIN_DOCUMENTS:1)(MAX_FEATURES:3)"))
R> summary(esa.mod2)
Call:
ore.odmESA(formula = ~TITLE, data = ESA_TEXT, odm.settings = list(case_id_column_name = "CUST_ID",
ESAS_MIN_ITEMS = 1), ctx.settings = list(TITLE = "TEXT(POLICY_NAME:ESA_TXTPOL)(TOKEN_TYPE:STEM)(MIN_DOCUMENTS:1)(MAX_FEATURES:3)"))
Settings:
value
min.items 1
topn.features 1000
value.threshold .00000001
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
odms.text.max.features 300000
odms.text.min.documents 3
prep.auto ON
Features:
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 TITLE.AIDS <NA> 1.0000000
2 2 TITLE.MARS <NA> 0.4078615
3 2 TITLE.ROVER <NA> 0.9130438
4 3 TITLE.MARS <NA> 1.0000000
5 4 TITLE.MARS <NA> 0.3011997
6 4 TITLE.NASA <NA> 0.6742695
7 4 TITLE.ROVER <NA> 0.6742695
8 5 TITLE.AIDS <NA> 1.0000000
9 6 TITLE.MARS <NA> 0.4078615
10 6 TITLE.NASA <NA> 0.9130438
11 7 TITLE.AIDS <NA> 1.0000000
R> settings(esa.mod2)
SETTING_NAME SETTING_VALUE SETTING_TYPE
1 ALGO_NAME ALGO_EXPLICIT_SEMANTIC_ANALYS INPUT
2 ESAS_MIN_ITEMS 1 INPUT
3 ESAS_TOPN_FEATURES 1000 DEFAULT
4 ESAS_VALUE_THRESHOLD .00000001 DEFAULT
5 ODMS_MISSING_VALUE_TREATMENT ODMS_MISSING_VALUE_AUTO DEFAULT
6 ODMS_SAMPLING ODMS_SAMPLING_DISABLE DEFAULT
7 ODMS_TEXT_MAX_FEATURES 300000 DEFAULT
8 ODMS_TEXT_MIN_DOCUMENTS 3 DEFAULT
9 PREP_AUTO ON INPUT
R> features(esa.mod2)
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 TITLE.AIDS <NA> 1.0000000
2 2 TITLE.MARS <NA> 0.4078615
3 2 TITLE.ROVER <NA> 0.9130438
4 3 TITLE.MARS <NA> 1.0000000
5 4 TITLE.MARS <NA> 0.3011997
6 4 TITLE.NASA <NA> 0.6742695
7 4 TITLE.ROVER <NA> 0.6742695
8 5 TITLE.AIDS <NA> 1.0000000
9 6 TITLE.MARS <NA> 0.4078615
10 6 TITLE.NASA <NA> 0.9130438
11 7 TITLE.AIDS <NA> 1.0000000
R> predict(esa.mod2, ESA_TEXT_CLOB, type = "class", supplemental.cols = "TITLE")
TITLE FEATURE_ID
1 Aids in Africa: Planning for a long war 1
2 Mars rover maneuvers for rim shot 2
3 Mars express confirms presence of water at Mars south pole 3
4 NASA announces major Mars rover finding 4
5 Drug access, Asia threat in focus at AIDS summit 1
6 NASA Mars Odyssey THEMIS image: typical crater 6
7 Road blocks for Aids 1
R>
R> ore.exec("Begin ctx_ddl.drop_policy('ESA_TXTPOL'); End;")
4.2.7 拡張可能Rアルゴリズム・モデルの構築
ore.odmRAlg関数は、OML4SQLを使用して拡張可能Rアルゴリズム・モデルを作成します。
拡張可能Rアルゴリズムは、登録済Rスクリプトを使用して、Rモデルを構築、スコア付けおよび表示します。また、分類、回帰、クラスタリング、特徴抽出、属性評価および相関機械学習機能をサポートします。
ore.odmRAlg関数の引数および関数の使用例の詳細は、help(ore.odmRAlg)をコールしてください。
拡張可能Rアルゴリズム・モデルの設定
次の表に、拡張可能Rアルゴリズム・モデルに適用される設定を示します。
表4-10 拡張可能Rアルゴリズム・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
Rアルゴリズムのマイニング・モデル作成ファンクションに対する、既存の登録済Rスクリプトの名前を指定します。Rスクリプトにより、トレーニング・データに対して最初に入力した引数のRファンクションが定義され、Rモデル・オブジェクトが戻されます。クラスタリングと特徴抽出のマイニング機能のモデル作成では、RモデルでR属性のdm$nclusおよびdm$nfeatを設定して、クラスタと特徴の数をそれぞれ指定する必要があります。
|
|
|
SELECT value param_name, ...FROM DUAL |
モデル作成ファンクションのオプションの入力パラメータに対する、数値および文字列のスカラーのリストを指定します。 |
|
|
|
既存の登録済Rスクリプトの名前を指定して、データをスコアリングします。スクリプトにより、対応する予測結果が含まれる |
|
|
|
スコアリングの各属性の重み(コントリビューション)を計算するRアルゴリズムに対する、既存の登録済Rスクリプトの名前を指定します。スクリプトにより、各属性の影響を与える重みが行内に含まれる |
RALG_DETAILS_FUNCTION |
R_DETAILS_FUNCTION_SCRIPT_NAME |
モデル情報を生成するRアルゴリズムに対する、既存の登録済Rスクリプトの名前を指定します。モデル・ビューの生成には、この設定が必要です。 |
RALG_DETAILS_FORMAT |
SELECT type_value column_name,FROM DUAL |
生成されるモデル・ビューの出力列タイプと列名の数値および文字列のスカラーのリストに対する、SELECT問合せを指定します。モデル・ビューの生成には、この設定が必要です。
|
例4-14 ore.odmRAlg関数の使用方法
library(OREembed)
digits <- getOption("digits")
options(digits = 5L)
IRIS <- ore.push(iris)
# Regression with glm
ore.scriptCreate("glm_build",
function(data, form, family)
glm(formula = form, data = data, family = family))
ore.scriptCreate("glm_score",
function(mod, data)
{ res <- predict(mod, newdata = data);
data.frame(res) })
ore.scriptCreate("glm_detail", function(mod)
data.frame(name=names(mod$coefficients),
coef=mod$coefficients))
ore.scriptList(name = "glm_build")
ore.scriptList(name = "glm_score")
ore.scriptList(name = "glm_detail")
ralg.glm <- ore.odmRAlg(IRIS, mining.function = "regression",
formula = c(form="Sepal.Length ~ ."),
build.function = "glm_build",
build.parameter = list(family="gaussian"),
score.function = "glm_score",
detail.function = "glm_detail",
detail.value = data.frame(name="a", coef=1))
summary(ralg.glm)
predict(ralg.glm, newdata = head(IRIS), supplemental.cols = "Sepal.Length")
ore.scriptDrop(name = "glm_build")
ore.scriptDrop(name = "glm_score")
ore.scriptDrop(name = "glm_detail")
# Classification with nnet
ore.scriptCreate("nnet_build",
function(dat, form, sz){
require(nnet);
set.seed(1234);
nnet(formula = formula(form), data = dat,
size = sz, linout = TRUE, trace = FALSE);
},
overwrite = TRUE)
ore.scriptCreate("nnet_detail", function(mod)
data.frame(conn = mod$conn, wts = mod$wts),
overwrite = TRUE)
ore.scriptCreate("nnet_score",
function(mod, data) {
require(nnet);
res <- data.frame(predict(mod, newdata = data));
names(res) <- sort(mod$lev); res
})
ralg.nnet <- ore.odmRAlg(IRIS, mining.function = "classification",
formula = c(form="Species ~ ."),
build.function = "nnet_build",
build.parameter = list(sz=2),
score.function = "nnet_score",
detail.function = "nnet_detail",
detail.value = data.frame(conn=1, wts =1))
summary(ralg.nnet)
predict(ralg.nnet, newdata = head(IRIS), supplemental.cols = "Species")
ore.scriptDrop(name = "nnet_build")
ore.scriptDrop(name = "nnet_score")
ore.scriptDrop(name = "nnet_detail")
# Feature extraction with pca
# Feature extraction with pca
ore.scriptCreate("pca_build",
function(dat){
mod <- prcomp(dat, retx = FALSE)
attr(mod, "dm$nfeat") <- ncol(mod$rotation)
mod},
overwrite = TRUE)
ore.scriptCreate("pca_score",
function(mod, data) {
res <- predict(mod, data)
as.data.frame(res)},
overwrite=TRUE)
ore.scriptCreate("pca_detail",
function(mod) {
rotation_t <- t(mod$rotation)
data.frame(id = seq_along(rownames(rotation_t)),
rotation_t)},
overwrite = TRUE)
X <- IRIS[, -5L]
ralg.pca <- ore.odmRAlg(X,
mining.function = "feature_extraction",
formula = NULL,
build.function = "pca_build",
score.function = "pca_score",
detail.function = "pca_detail",
detail.value = data.frame(Feature.ID=1,
ore.pull(head(X,1L))))
summary(ralg.pca)
head(cbind(X, Pred = predict(ralg.pca, newdata = X)))
ore.scriptDrop(name = "pca_build")
ore.scriptDrop(name = "pca_score")
ore.scriptDrop(name = "pca_detail")
options(digits = digits)この例のリスト
R> library(OREembed)
R>
R> digits <- getOption("digits")
R> options(digits = 5L)
R>
R> IRIS <- ore.push(iris)
R>
R> # Regression with glm
R> ore.scriptCreate("glm_build",
+ function(data, form, family)
+ glm(formula = form, data = data, family = family))
R>
R> ore.scriptCreate("glm_score",
+ function(mod, data)
+ { res <- predict(mod, newdata = data);
+ data.frame(res) })
R>
R> ore.scriptCreate("glm_detail", function(mod)
+ data.frame(name=names(mod$coefficients),
+ coef=mod$coefficients))
R>
R> ore.scriptList(name = "glm_build")
NAME SCRIPT
1 glm_build function (data, form, family) \nglm(formula = form, data = data, family = family)
R> ore.scriptList(name = "glm_score")
NAME SCRIPT
1 glm_score function (mod, data) \n{\n res <- predict(mod, newdata = data)\n data.frame(res)\n}
R> ore.scriptList(name = "glm_detail")
NAME SCRIPT
1 glm_detail function (mod) \ndata.frame(name = names(mod$coefficients), coef = mod$coefficients)
R>
R> ralg.glm <- ore.odmRAlg(IRIS, mining.function = "regression",
+ formula = c(form="Sepal.Length ~ ."),
+ build.function = "glm_build",
+ build.parameter = list(family="gaussian"),
+ score.function = "glm_score",
+ detail.function = "glm_detail",
+ detail.value = data.frame(name="a", coef=1))
R>
R> summary(ralg.glm)
Call:
ore.odmRAlg(data = IRIS, mining.function = "regression", formula = c(form = "Sepal.Length ~ ."),
build.function = "glm_build", build.parameter = list(family = "gaussian"),
score.function = "glm_score", detail.function = "glm_detail",
detail.value = data.frame(name = "a", coef = 1))
Settings:
value
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto OFF
build.function OML_USER.glm_build
build.parameter select 'Sepal.Length ~ .' "form", 'gaussian' "family" from dual
details.format select cast('a' as varchar2(4000)) "name", 1 "coef" from dual
details.function OML_USER.glm_detail
score.function OML_USER.glm_score
name coef
1 (Intercept) 2.17127
2 Petal.Length 0.82924
3 Petal.Width -0.31516
4 Sepal.Width 0.49589
5 Speciesversicolor -0.72356
6 Speciesvirginica -1.02350
R> predict(ralg.glm, newdata = head(IRIS), supplemental.cols = "Sepal.Length")
Sepal.Length PREDICTION
1 5.1 5.0048
2 4.9 4.7568
3 4.7 4.7731
4 4.6 4.8894
5 5.0 5.0544
6 5.4 5.3889
R>
R> ore.scriptDrop(name = "glm_build")
R> ore.scriptDrop(name = "glm_score")
R> ore.scriptDrop(name = "glm_detail")
R>
R> # Classification with nnet
R> ore.scriptCreate("nnet_build",
+ function(dat, form, sz){
+ require(nnet);
+ set.seed(1234);
+ nnet(formula = formula(form), data = dat,
+ size = sz, linout = TRUE, trace = FALSE);
+ },
+ overwrite = TRUE)
R>
R> ore.scriptCreate("nnet_detail", function(mod)
+ data.frame(conn = mod$conn, wts = mod$wts),
+ overwrite = TRUE)
R>
R> ore.scriptCreate("nnet_score",
+ function(mod, data) {
+ require(nnet);
+ res <- data.frame(predict(mod, newdata = data));
+ names(res) <- sort(mod$lev); res
+ })
R>
R> ralg.nnet <- ore.odmRAlg(IRIS, mining.function = "classification",
+ formula = c(form="Species ~ ."),
+ build.function = "nnet_build",
+ build.parameter = list(sz=2),
+ score.function = "nnet_score",
+ detail.function = "nnet_detail",
+ detail.value = data.frame(conn=1, wts =1))
R>
R> summary(ralg.nnet)
Call:
ore.odmRAlg(data = IRIS, mining.function = "classification",
formula = c(form = "Species ~ ."), build.function = "nnet_build",
build.parameter = list(sz = 2), score.function = "nnet_score",
detail.function = "nnet_detail", detail.value = data.frame(conn = 1,
wts = 1))
Settings:
value
clas.weights.balanced OFF
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto OFF
build.function OML_USER.nnet_build
build.parameter select 'Species ~ .' "form", 2 "sz" from dual
details.format select 1 "conn", 1 "wts" from dual
details.function OML_USER.nnet_detail
score.function OML_USER.nnet_score
conn wts
1 0 1.46775
2 1 -12.88542
3 2 -4.38886
4 3 9.98648
5 4 16.57056
6 0 0.97809
7 1 -0.51626
8 2 -0.94815
9 3 0.13692
10 4 0.35104
11 0 37.22475
12 5 -66.49123
13 6 70.81160
14 0 -4.50893
15 5 7.01611
16 6 20.88774
17 0 -32.15127
18 5 58.92088
19 6 -91.96989
R> predict(ralg.nnet, newdata = head(IRIS), supplemental.cols = "Species")
Species PREDICTION PROBABILITY
1 setosa setosa 0.99999
2 setosa setosa 0.99998
3 setosa setosa 0.99999
4 setosa setosa 0.99998
5 setosa setosa 1.00000
6 setosa setosa 0.99999
R>
R> ore.scriptDrop(name = "nnet_build")
R> ore.scriptDrop(name = "nnet_score")
R> ore.scriptDrop(name = "nnet_detail")
R>
R> ore.scriptCreate("pca_build",
+ function(dat){
+ mod <- prcomp(dat, retx = FALSE)
+ attr(mod, "dm$nfeat") <- ncol(mod$rotation)
+ mod},
+ overwrite = TRUE)
R>
R> ore.scriptCreate("pca_score",
+ function(mod, data) {
+ res <- predict(mod, data)
+ as.data.frame(res)},
+ overwrite=TRUE)
R>
R> ore.scriptCreate("pca_detail",
+ function(mod) {
+ rotation_t <- t(mod$rotation)
+ data.frame(id = seq_along(rownames(rotation_t)),
+ rotation_t)},
+ overwrite = TRUE)
R>
R> X <- IRIS[, -5L]
R> ralg.pca <- ore.odmRAlg(X,
+ mining.function = "feature_extraction",
+ formula = NULL,
+ build.function = "pca_build",
+ score.function = "pca_score",
+ detail.function = "pca_detail",
+ detail.value = data.frame(Feature.ID=1,
+ ore.pull(head(X,1L))))
R>
R> summary(ralg.pca)
Call:
ore.odmRAlg(data = X, mining.function = "feature_extraction",
formula = NULL, build.function = "pca_build", score.function = "pca_score",
detail.function = "pca_detail", detail.value = data.frame(Feature.ID = 1,
ore.pull(head(X, 1L))))
Settings:
value
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto OFF
build.function OML_USER.pca_build
details.format select 1 "Feature.ID", 5.1 "Sepal.Length", 3.5 "Sepal.Width", 1.4 "Petal.Length", 0.2 "Petal.Width" from dual
details.function OML_USER.pca_detail
score.function OML_USER.pca_score
Feature.ID Sepal.Length Sepal.Width Petal.Length Petal.Width
1 1 0.856671 0.358289 0.36139 -0.084523
2 2 -0.173373 -0.075481 0.65659 0.730161
3 3 0.076236 0.545831 -0.58203 0.597911
4 4 0.479839 -0.753657 -0.31549 0.319723
R> head(cbind(X, Pred = predict(ralg.pca, newdata = X)))
Sepal.Length Sepal.Width Petal.Length Petal.Width FEATURE_ID
1 5.1 3.5 1.4 0.2 2
2 4.9 3.0 1.4 0.2 4
3 4.7 3.2 1.3 0.2 3
4 4.6 3.1 1.5 0.2 4
5 5.0 3.6 1.4 0.2 2
6 5.4 3.9 1.7 0.4 2
R>
R> ore.scriptDrop(name = "pca_build")
R> ore.scriptDrop(name = "pca_score")
R> ore.scriptDrop(name = "pca_detail")
R>
R> options(digits = digits)4.2.8 一般化線形モデル
ore.odmGLM関数は一般化線形モデル(GLM)を構築しますが、これは線形モデル(線形回帰)のクラスを含み、このクラスを拡張したものです。
一般化線形モデルは、実際には違反されることが多い線形モデルの制限を緩和したものです。たとえば、2値(yes/noまたは0/1)応答は、クラス間で同じ分散を持ちません。
OML4SQLのGLMはパラメトリックなモデリング手法です。パラメトリック・モデルでは、データの分散を仮定します。仮定が満たされる場合、パラメトリック・モデルはノンパラメトリック・モデルよりも効率的になります。
このタイプのモデルの作成では、どの程度仮定が満たされるかを見極めることが課題となります。このため、良質なパラメトリック・モデルを作成するには質の診断が重要です。
古典的な、線形回帰における重み付き最小二乗推定およびロジスティック回帰における反復再重み付き最小二乗推定(いずれもコレスキー分解およびマトリクス反転を使用する解決)に加えて、OML4SQLのGLMでは、マトリクス反転が不要で高次元データに最適な共役勾配法に基づく最適化アルゴリズムを提供します。アルゴリズムの選択は内部的に処理され、ユーザーに対して透過的です。
GLMを使用して、次のような分類モデルまたは回帰モデルを構築できます。
-
分類: 2項ロジスティック回帰は、GLM分類アルゴリズムです。このアルゴリズムでは、ロジット・リンク関数および2項分散関数を使用します。
-
回帰: 線形回帰は、GLM回帰アルゴリズムです。このアルゴリズムでは、ターゲット値の範囲に対する一定分散およびターゲット変換を想定していません。
ore.odmGLM関数では、2つの異なるタイプのモデルを構築できます。一部、分類モデルにのみ適用される引数、回帰モデルにのみ適用される引数があります。
ore.odmGLM関数の引数の詳細は、help(ore.odmGLM)を呼び出してください。
次の例では、GLMを使用していくつかのモデルを構築します。入力ore.frameオブジェクトは、データベースにプッシュされるRデータセットです。
一般化線形モデルの設定
次の表に、一般化線形モデルに適用される設定を示します。
表4-11 一般化線形モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
信頼区間の信頼水準。 デフォルトの信頼水準は |
|
|
|
特徴生成が二次式または三次式のどちらであるかを示します。 特徴の生成を有効にすると、アルゴリズムでは、最適な特徴生成方式がデータに基づいて自動的に選択されます。 |
|
|
|
GLMに対して特徴生成を有効にするかどうかの指定。デフォルトでは、特徴生成は有効化されていません。 ノート: 特徴生成は、特徴選択が有効な場合にのみ有効です。 |
|
|
|
モデルに特徴を追加する際の、特徴選択のペナルティ基準。 特徴選択を有効にすると、アルゴリズムでは、ペナルティ基準がデータに基づいて自動的に選択されます。 |
|
|
|
GLMに対して特徴選択を有効にするかどうかの指定。 デフォルトでは、特徴選択は無効です。 |
|
|
|
特徴選択を有効にすると、この設定では、最終モデルで選択可能な特徴の最大数が指定されます。 デフォルトでは、十分なメモリーを確保するために、アルゴリズムによって特徴の数が制限されます。 |
GLMS_PRUNE_MODEL |
|
最終モデルでの特徴のプルーニングを有効また無効にします。プルーニングは、線形回帰の場合はt検定統計に、ロジスティック回帰の場合はWald検定統計に基づきます。すべての特徴が全データに対して統計的な意味付けを持つまで、これらの特徴はループ処理でプルーニングされます。 特徴選択を有効にすると、アルゴリズムでは、プルーニングがデータに基づいて自動的に実行されます。 |
|
|
target_value |
バイナリ・ロジスティック回帰モデルで参照クラスとして使用されるターゲット値。非参照クラスに対しては確率が生成されます。 デフォルトでは、参照クラスの場合、最も普及率の高い値(ほとんどのケース)がアルゴリズムによって選択されます。 |
GLMS_RIDGE_REGRESSION |
|
リッジ回帰を有効または無効にします。リッジは、回帰と分類の両方のマイニング機能に適用されます。 リッジを有効にすると、 ノート: リッジは、特徴選択が指定されていない場合または明示的に無効にされている場合にのみ有効です。リッジ回帰と特徴選択を両方とも明示的に有効にすると、例外が発生します。 |
|
|
|
リッジ・パラメータ値。この設定は、アルゴリズムでリッジ回帰の使用が構成されている場合にのみ使用されます。 リッジ回帰がアルゴリズムによって内部的に有効になっている場合、リッジ・パラメータはアルゴリズムによって決定されます。 |
|
|
|
行診断を有効または無効にします。 |
|
|
範囲は( |
GLMアルゴリズムの収束許容値の設定 デフォルト値は、システムによって決定されます。 |
|
|
正の整数 |
GLMアルゴリズムの最大反復数。デフォルト値は、システムによって決定されます。 |
|
|
0または正の整数
|
SGDソルバーで使用されるバッチ内の行数。このパラメータの値によって、SGDソルバーのバッチのサイズが設定されます。0を入力すると、データ駆動のバッチ・サイズの推定値がトリガーされます。 デフォルトは |
|
|
|
この設定により、ユーザーがGLMソルバーを選択できます。 |
|
|
|
この設定により、ユーザーはスパース・ソルバーを使用できます(使用可能な場合)。デフォルト値は |
例4-15 線形回帰モデルの構築
この例では、longleyデータセットを使用して線形回帰モデルを構築します。
longley_of <- ore.push(longley) longfit1 <- ore.odmGLM(Employed ~ ., data = longley_of) summary(longfit1)
この例のリスト
R> longley_of <- ore.push(longley)
R> longfit1 <- ore.odmGLM(Employed ~ ., data = longley_of)
R> summary(longfit1)
Call:
ore.odmGLM(formula = Employed ~ ., data = longely_of)
Residuals:
Min 1Q Median 3Q Max
-0.41011 -0.15767 -0.02816 0.10155 0.45539
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -3.482e+03 8.904e+02 -3.911 0.003560 **
GNP.deflator 1.506e-02 8.492e-02 0.177 0.863141
GNP -3.582e-02 3.349e-02 -1.070 0.312681
Unemployed -2.020e-02 4.884e-03 -4.136 0.002535 **
Armed.Forces -1.033e-02 2.143e-03 -4.822 0.000944 ***
Population -5.110e-02 2.261e-01 -0.226 0.826212
Year 1.829e+00 4.555e-01 4.016 0.003037 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.3049 on 9 degrees of freedom
Multiple R-squared: 0.9955, Adjusted R-squared: 0.9925
F-statistic: 330.3 on 6 and 9 DF, p-value: 4.984e-10
例4-16 ore.odmGLMモデルの係数に対するリッジ推定の使用方法
この例では、前の例のlongley_ofのore.frameを使用します。この例では、ore.odmGLM関数を呼び出し、係数のリッジ推定を使用して指定します。
longfit2 <- ore.odmGLM(Employed ~ ., data = longley_of, ridge = TRUE,
ridge.vif = TRUE)
summary(longfit2)この例のリスト
R> longfit2 <- ore.odmGLM(Employed ~ ., data = longley_of, ridge = TRUE,
+ ridge.vif = TRUE)
R> summary(longfit2)
Call:
ore.odmGLM(formula = Employed ~ ., data = longley_of, ridge = TRUE,
ridge.vif = TRUE)
Residuals:
Min 1Q Median 3Q Max
-0.4100 -0.1579 -0.0271 0.1017 0.4575
Coefficients:
Estimate VIF
(Intercept) -3.466e+03 0.000
GNP.deflator 1.479e-02 0.077
GNP -3.535e-02 0.012
Unemployed -2.013e-02 0.000
Armed.Forces -1.031e-02 0.000
Population -5.262e-02 0.548
Year 1.821e+00 2.212
Residual standard error: 0.3049 on 9 degrees of freedom
Multiple R-squared: 0.9955, Adjusted R-squared: 0.9925
F-statistic: 330.2 on 6 and 9 DF, p-value: 4.986e-10
例4-17 ロジスティック回帰GLMの構築
この例では、ロジスティック回帰(分類)モデルを構築します。ここでは、infertデータセットを使用します。この例では、ore.odmGLM関数を呼び出し、logisticをtype引数(二項分布GLMを構築します)として指定します。
infert_of <- ore.push(infert)
infit1 <- ore.odmGLM(case ~ age+parity+education+spontaneous+induced,
data = infert_of, type = "logistic")
infit1この例のリスト
R> infert_of <- ore.push(infert)
R> infit1 <- ore.odmGLM(case ~ age+parity+education+spontaneous+induced,
+ data = infert_of, type = "logistic")
R> infit1
Response:
case == "1"
Call: ore.odmGLM(formula = case ~ age + parity + education + spontaneous +
induced, data = infert_of, type = "logistic")
Coefficients:
(Intercept) age parity education0-5yrs education12+ yrs spontaneous induced
-2.19348 0.03958 -0.82828 1.04424 -0.35896 2.04590 1.28876
Degrees of Freedom: 247 Total (i.e. Null); 241 Residual
Null Deviance: 316.2
Residual Deviance: 257.8 AIC: 271.8
例4-18 ロジスティック回帰GLMの構築での参照値の指定
この例では、ロジスティック回帰(分類)モデルを構築し、参照値を指定します。この例では、例4-17のinfert_ofのore.frameを使用します。
infit2 <- ore.odmGLM(case ~ age+parity+education+spontaneous+induced,
data = infert_of, type = "logistic", reference = 1)
infit2この例のリスト
infit2 <- ore.odmGLM(case ~ age+parity+education+spontaneous+induced,
data = infert_of, type = "logistic", reference = 1)
infit2
Response:
case == "0"
Call: ore.odmGLM(formula = case ~ age + parity + education + spontaneous +
induced, data = infert_of, type = "logistic", reference = 1)
Coefficients:
(Intercept) age parity education0-5yrs education12+ yrs spontaneous induced
2.19348 -0.03958 0.82828 -1.04424 0.35896 -2.04590 -1.28876
Degrees of Freedom: 247 Total (i.e. Null); 241 Residual
Null Deviance: 316.2
Residual Deviance: 257.8 AIC: 271.84.2.9 k-Means
ore.odmKM関数はOML4SQLのk-Means (KM)アルゴリズムを使用します。このアルゴリズムは、指定した数のクラスタにデータをパーティション化する、距離ベースのクラスタリング・アルゴリズムです。
このアルゴリズムには、次の機能があります。
-
複数の距離関数(ユークリッド、コサインおよび高速コサインの各距離関数)。デフォルトはユークリッドです。
-
クラスタごとに、アルゴリズムによって、重心、各属性のヒストグラム、およびクラスタに割り当てられるデータの大部分を囲むハイパーボックスを記述するルールが戻されます。重心は、カテゴリ属性については最頻値を、数値属性については平均および分散をレポートします。
ore.odmKM関数の引数の詳細は、help(ore.odmKM)をコールしてください。
k-Meansモデルの設定
次の表に、k-Meansモデルに適用される設定を示します。
表4-12 k-Meansモデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
k-Meansの最小収束許容値。最小収束許容値を満たすか、 収束許容値が減少すると、ソリューションはより正確になりますが、実行時間が長くなることがあります。 デフォルトの収束許容値は、 |
|
|
|
k-Meansの距離関数。 デフォルトの距離ファンクションは |
|
|
|
k-Meansの最大反復数。最大反復数に到達するか、 デフォルトの反復回数は |
|
|
|
クラスタのルール記述にその属性を含めるために必要な、非NULL値でなければならない属性値の最小パーセント。 データがまばらであったり多くの欠損値を含んでいる場合、最小支持度が高すぎると非常に短いルールまたは空のルールになる可能性があります。 デフォルトの最小支持度は、 |
|
|
|
k-Meansで生成される属性ヒストグラムでのビンの数。各属性のビン境界は、トレーニング・データセット全体でグローバルに計算されます。ビニングの方法は等幅です。ただ1つのビンを持つ単一値が含まれる属性を除き、すべての属性は同じ数のビンを持ちます。 デフォルトのヒストグラム・ビン数は |
|
|
|
k-Meansの分割基準。分割基準は、新しいk-Meansクラスタの初期化を制御します。アルゴリズムがバイナリ・ツリーを作成し、同時に新しいクラスタを1つ追加します。 分割基準がサイズに基づいている場合、新しいクラスタは現在最も大きなクラスタのある領域に置かれます。分割基準が分散値に基づいている場合、新しいクラスタは最も拡大されたクラスタの領域に置かれます。 デフォルトの分割基準は |
KMNS_RANDOM_SEED |
負でない整数 |
この設定により、k-Meansの初期化中に使用されるランダム・ジェネレータのシードを制御します。これは、負でない整数である必要があります。 デフォルトは |
|
|
|
この設定により、作成中に計算されるクラスタ詳細のレベルが決まります。
|
例4-19 ore.odmKM関数の使用方法
この例では、ore.odmKMeans関数の使用方法を示します。この例では、100行および2列を含むマトリクスを2つ作成します。行の値は無作為変量です。それらのマトリクスをmatrix xにバインドした後、xをdata.frameに強制変換し、それをore.frameオブジェクトであるx_ofとしてデータベースにプッシュします。この例では次に、ore.odmKMeans関数をコールして、KMモデルのkm.mod1を構築します。その後、summary関数およびhistogram関数をそのモデルでコールします。図4-2に、histogram関数で表示されるグラフを示します。
この例では最後に、そのモデルを使用して予測を行い、結果をローカル・メモリーにプルして、結果を表示します。図4-3に、points関数で表示されるグラフを示します。
x <- rbind(matrix(rnorm(100, sd = 0.3), ncol = 2),
matrix(rnorm(100, mean = 1, sd = 0.3), ncol = 2))
colnames(x) <- c("x", "y")
x_of <- ore.push (data.frame(x))
km.mod1 <- NULL
km.mod1 <- ore.odmKMeans(~., x_of, num.centers=2)
summary(km.mod1)
histogram(km.mod1)
# Make a prediction.
km.res1 <- predict(km.mod1, x_of, type="class", supplemental.cols=c("x","y"))
head(km.res1, 3)
# Pull the results to the local memory and plot them.
km.res1.local <- ore.pull(km.res1)
plot(data.frame(x=km.res1.local$x, y=km.res1.local$y),
col=km.res1.local$CLUSTER_ID)
points(km.mod1$centers2, col = rownames(km.mod1$centers2), pch = 8, cex=2)
head(predict(km.mod1, x_of, type=c("class","raw"),
supplemental.cols=c("x","y")), 3)この例のリストR> x <- rbind(matrix(rnorm(100, sd = 0.3), ncol = 2),
+ matrix(rnorm(100, mean = 1, sd = 0.3), ncol = 2))
R> colnames(x) <- c("x", "y")
R> x_of <- ore.push (data.frame(x))
R> km.mod1 <- NULL
R> km.mod1 <- ore.odmKMeans(~., x_of, num.centers=2)
R> summary(km.mod1)
Call:
ore.odmKMeans(formula = ~., data = x_of, num.centers = 2)
Settings:
value
clus.num.clusters 2
block.growth 2
conv.tolerance 0.01
distance euclidean
iterations 3
min.pct.attr.support 0.1
num.bins 10
split.criterion variance
prep.auto on
Centers:
x y
2 0.99772307 0.93368684
3 -0.02721078 -0.05099784
R> histogram(km.mod1)
R> # Make a prediction.
R> km.res1 <- predict(km.mod1, x_of, type="class", supplemental.cols=c("x","y"))
R> head(km.res1, 3)
x y CLUSTER_ID
1 -0.03038444 0.4395409 3
2 0.17724606 -0.5342975 3
3 -0.17565761 0.2832132 3
# Pull the results to the local memory and plot them.
R> km.res1.local <- ore.pull(km.res1)
R> plot(data.frame(x=km.res1.local$x, y=km.res1.local$y),
+ col=km.res1.local$CLUSTER_ID)
R> points(km.mod1$centers2, col = rownames(km.mod1$centers2), pch = 8, cex=2)
R> head(predict(km.mod1, x_of, type=c("class","raw"),
supplemental.cols=c("x","y")), 3)
'2' '3' x y CLUSTER_ID
1 8.610341e-03 0.9913897 -0.03038444 0.4395409 3
2 8.017890e-06 0.9999920 0.17724606 -0.5342975 3
3 5.494263e-04 0.9994506 -0.17565761 0.2832132 3
4.2.10 Naive Bayes
ore.odmNB関数は、OML4SQLのNaive Bayesモデルを構築します。
Naive Bayesアルゴリズムは条件付き確率に基づいています。Naive Bayesは履歴データを検索し、属性値の頻度と属性値の組合せの頻度を観測することによってターゲット値の条件付き確率を計算します。
Naive Bayesでは、各予測子は他の予測子とは条件的に独立していると想定されます。(Bayesの定理では、予測子が独立している必要があります。)
ore.odmNB関数の引数の詳細は、help(ore.odmNB)をコールしてください。
Naive Bayesモデルの設定
次の表に、Naive Bayesモデルに適用される設定を示します。
表4-13 Naive Bayesモデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
NBアルゴリズムでの組しきい値。 デフォルトは |
|
|
|
NBアルゴリズムでの単一しきい値。 デフォルト値は |
例4-20 ore.odmNB関数の使用方法
この例では、入力ore.frameを作成し、Naive Bayesモデルを構築して、予測を行い、混同マトリクスを生成します。
m <- mtcars m$gear <- as.factor(m$gear) m$cyl <- as.factor(m$cyl) m$vs <- as.factor(m$vs) m$ID <- 1:nrow(m) mtcars_of <- ore.push(m) row.names(mtcars_of) <- mtcars_of # Build the model. nb.mod <- ore.odmNB(gear ~ ., mtcars_of) summary(nb.mod) # Make predictions and generate a confusion matrix. nb.res <- predict (nb.mod, mtcars_of, "gear") with(nb.res, table(gear, PREDICTION))
この例のリスト
R> m <- mtcars
R> m$gear <- as.factor(m$gear)
R> m$cyl <- as.factor(m$cyl)
R> m$vs <- as.factor(m$vs)
R> m$ID <- 1:nrow(m)
R> mtcars_of <- ore.push(m)
R> row.names(mtcars_of) <- mtcars_of
R> # Build the model.
R> nb.mod <- ore.odmNB(gear ~ ., mtcars_of)
R> summary(nb.mod)
Call:
ore.odmNB(formula = gear ~ ., data = mtcars_of)
Settings:
value
prep.auto on
Apriori:
3 4 5
0.46875 0.37500 0.15625
Tables:
$ID
( ; 26.5), [26.5; 26.5] (26.5; )
3 1.00000000
4 0.91666667 0.08333333
5 1.00000000
$am
0 1
3 1.0000000
4 0.3333333 0.6666667
5 1.0000000
$cyl
'4', '6' '8'
3 0.2 0.8
4 1.0
5 0.6 0.4
$disp
( ; 196.299999999999995), [196.299999999999995; 196.299999999999995]
3 0.06666667
4 1.00000000
5 0.60000000
(196.299999999999995; )
3 0.93333333
4
5 0.40000000
$drat
( ; 3.385), [3.385; 3.385] (3.385; )
3 0.8666667 0.1333333
4 1.0000000
5 1.0000000
$hp
( ; 136.5), [136.5; 136.5] (136.5; )
3 0.2 0.8
4 1.0
5 0.4 0.6
$vs
0 1
3 0.8000000 0.2000000
4 0.1666667 0.8333333
5 0.8000000 0.2000000
$wt
( ; 3.2024999999999999), [3.2024999999999999; 3.2024999999999999]
3 0.06666667
4 0.83333333
5 0.80000000
(3.2024999999999999; )
3 0.93333333
4 0.16666667
5 0.20000000
Levels:
[1] "3" "4" "5"
R> # Make predictions and generate a confusion matrix.
R> nb.res <- predict (nb.mod, mtcars_of, "gear")
R> with(nb.res, table(gear, PREDICTION))
PREDICTION
gear 3 4 5
3 14 1 0
4 0 12 0
5 0 1 44.2.11 Non-Negative Matrix Factorization
ore.odmNMF関数は、特徴抽出用にOML4SQLのNon-Negative Matrix Factorization (NMF)モデルを構築します。
NMFによって抽出される各特徴は、元の属性セットの線形結合です。各特徴には、負でない一連の係数があり、それらは特徴の各属性の重みのメジャーです。引数allow.negative.scoresがTRUEの場合、負の係数が許可されます。
ore.odmNMF関数の引数の詳細は、help(ore.odmNMF)をコールしてください。
Non-Negative Matrix Factorizationモデルの設定
次の表に、Non-Negative Matrix Factorizationモデルに適用される設定を示します。
表4-14 Non-Negative Matrix Factorizationモデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
NMFアルゴリズムでの収束許容値。 デフォルトは |
|
|
|
スコアリング結果で負数を許可するかどうか。 デフォルトは |
|
|
|
NMFアルゴリズムの反復回数。 デフォルトは |
|
|
|
NMFアルゴリズムのランダム・シード。 デフォルトは |
例4-21 ore.odmNMF関数の使用方法
この例では、トレーニング・データセットに基づいてNMFモデルを作成し、テスト・データセットに基づいてスコアリングします。
training.set <- ore.push(npk[1:18, c("N","P","K")])
scoring.set <- ore.push(npk[19:24, c("N","P","K")])
nmf.mod <- ore.odmNMF(~., training.set, num.features = 3)
features(nmf.mod)
summary(nmf.mod)
predict(nmf.mod, scoring.set)この例のリスト
R> training.set <- ore.push(npk[1:18, c("N","P","K")])
R> scoring.set <- ore.push(npk[19:24, c("N","P","K")])
R> nmf.mod <- ore.odmNMF(~., training.set, num.features = 3)
R> features(nmf.mod)
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 K 0 3.723468e-01
2 1 K 1 1.761670e-01
3 1 N 0 7.469067e-01
4 1 N 1 1.085058e-02
5 1 P 0 5.730082e-01
6 1 P 1 2.797865e-02
7 2 K 0 4.107375e-01
8 2 K 1 2.193757e-01
9 2 N 0 8.065393e-03
10 2 N 1 8.569538e-01
11 2 P 0 4.005661e-01
12 2 P 1 4.124996e-02
13 3 K 0 1.918852e-01
14 3 K 1 3.311137e-01
15 3 N 0 1.547561e-01
16 3 N 1 1.283887e-01
17 3 P 0 9.791965e-06
18 3 P 1 9.113922e-01
R> summary(nmf.mod)
Call:
ore.odmNMF(formula = ~., data = training.set, num.features = 3)
Settings:
value
feat.num.features 3
nmfs.conv.tolerance .05
nmfs.nonnegative.scoring nmfs.nonneg.scoring.enable
nmfs.num.iterations 50
nmfs.random.seed -1
prep.auto on
Features:
FEATURE_ID ATTRIBUTE_NAME ATTRIBUTE_VALUE COEFFICIENT
1 1 K 0 3.723468e-01
2 1 K 1 1.761670e-01
3 1 N 0 7.469067e-01
4 1 N 1 1.085058e-02
5 1 P 0 5.730082e-01
6 1 P 1 2.797865e-02
7 2 K 0 4.107375e-01
8 2 K 1 2.193757e-01
9 2 N 0 8.065393e-03
10 2 N 1 8.569538e-01
11 2 P 0 4.005661e-01
12 2 P 1 4.124996e-02
13 3 K 0 1.918852e-01
14 3 K 1 3.311137e-01
15 3 N 0 1.547561e-01
16 3 N 1 1.283887e-01
17 3 P 0 9.791965e-06
18 3 P 1 9.113922e-01
R> predict(nmf.mod, scoring.set)
'1' '2' '3' FEATURE_ID
19 0.1972489 1.2400782 0.03280919 2
20 0.7298919 0.0000000 1.29438165 3
21 0.1972489 1.2400782 0.03280919 2
22 0.0000000 1.0231268 0.98567623 2
23 0.7298919 0.0000000 1.29438165 3
24 1.5703239 0.1523159 0.00000000 14.2.12 直交パーティショニング・クラスタ
ore.odmOC関数は、直交パーティショニング・クラスタ(O-Cluster)アルゴリズムを使用してOML4SQLモデルを構築します。
O-Clusterアルゴリズムは、グリッドベースの階層クラスタリング・モデルを構築しますが、つまり、軸並行な(直行の)パーティションを入力属性空間に作成します。このアルゴリズムは再帰的に作用します。生成される階層構造は、属性空間をクラスタに分割する不規則なグリッドになります。生成されるクラスタは、属性空間内の密度の高い領域を定義します。
クラスタは、属性軸沿いの間隔と、対応する重心およびヒストグラムによって記述されます。sensitivity引数は、基準となる密度レベルを定義します。最大密度がこの基準水準を上回る領域のみがクラスタとして認識されます。
k-Meansアルゴリズムの場合、自然なクラスタが存在しない可能性があっても、空間を分割します。たとえば、密度が均一な領域がある場合、k-Meansは、その領域をn個のクラスタ(nはユーザーが指定します)に分割します。O-Clusterは、低密度の領域に切断面を配置して、高密度の領域を切り離します。O-Clusterでは、複数の最頻値を持つヒストグラム(ピークと谷)を必要とします。ある領域に、密度が均一または単調に変化している投影がある場合、O-Clusterはその領域をパーティション化しません。
O-Clusterによって発見されたクラスタを使用して、ベイズ確率モデルが生成され、このモデルがpredict関数によるスコアリングの際に使用され、データ・ポイントをクラスタに割り当てます。生成される確率モデルは混合モデルで、混合要素は、量的属性の独立正規分布と質的属性の多項分布の積によって表されます。
O-Cluster用のデータをユーザー自身が準備する場合は、次の点に留意してください。
-
O-Clusterアルゴリズムでは、モデルを構築する際に、必ずしもすべての入力データを使用するわけではありません。データはバッチで読み込みます(デフォルトのバッチ・サイズは50,000)。統計テストに基づいて、まだ見つかっていないクラスタが存在すると考えられる場合にのみ、別のバッチを読み込みます。
-
O-Clusterは、すべてのデータを読み込むことなくモデルの作成を停止する場合があるので、データをランダム化しておくことをお薦めします。
-
2項属性は、質的属性として宣言する必要があります。O-Clusterは、質的データを数値にマップします。
-
ビンの必要数の概算が自動化されたOML4SQLの等幅ビニング変換を使用することをお薦めします。
-
外れ値が存在すると、クラスタリング・アルゴリズムに大きな影響を与える可能性があります。ビニングまたは正規化を行う前に、クリッピング変換を実行してください。等幅ビニングで外れ値が存在すると、O-Clusterでクラスタを検出できなくなる場合があります。その結果、母集団全体が1つのクラスタ内に含まれているように見えます。
formula引数の仕様には~ termsがあり、ここでtermsはモデルに含まれる列名です。複数のterms項目は列名の間に+を使用することで指定されます。~ .は、dataのすべての列をモデル構築に使用する場合に使用します。列を除外するには、除外する各列名の前に-を使用します。
ore.odmOC関数の引数の詳細は、help(ore.odmOC)をコールしてください。
直交パーティショニング・クラスタ・モデルの設定
次の表に、直交パーティショニング・クラスタ・モデルに適用される設定を示します。
表4-15 直交パーティショニング・クラスタ・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
新しいクラスタを分割するために必要な最大密度を指定する割合。この割合は、全体の均一密度と関連しています。 デフォルトは |
例4-22 ore.odmOC関数の使用方法
この例では、統合データセットに基づいてO-Clusterモデルを作成します。例の後の図に、生成されるクラスタのヒストグラムを示します。
x <- rbind(matrix(rnorm(100, mean = 4, sd = 0.3), ncol = 2),
matrix(rnorm(100, mean = 2, sd = 0.3), ncol = 2))
colnames(x) <- c("x", "y")
x_of <- ore.push (data.frame(ID=1:100,x))
rownames(x_of) <- x_of$ID
oc.mod <- ore.odmOC(~., x_of, num.centers=2)
summary(oc.mod)
histogram(oc.mod)
predict(oc.mod, x_of, type=c("class","raw"), supplemental.cols=c("x","y"))この例のリスト
R> x <- rbind(matrix(rnorm(100, mean = 4, sd = 0.3), ncol = 2),
+ matrix(rnorm(100, mean = 2, sd = 0.3), ncol = 2))
R> colnames(x) <- c("x", "y")
R> x_of <- ore.push (data.frame(ID=1:100,x))
R> rownames(x_of) <- x_of$ID
R> oc.mod <- ore.odmOC(~., x_of, num.centers=2)
R> summary(oc.mod)
Call:
ore.odmOC(formula = ~., data = x_of, num.centers = 2)
Settings:
value
clus.num.clusters 2
max.buffer 50000
sensitivity 0.5
prep.auto on
Clusters:
CLUSTER_ID ROW_CNT PARENT_CLUSTER_ID TREE_LEVEL DISPERSION IS_LEAF
1 1 100 NA 1 NA FALSE
2 2 56 1 2 NA TRUE
3 3 43 1 2 NA TRUE
Centers:
MEAN.x MEAN.y
2 1.85444 1.941195
3 4.04511 4.111740
R> histogram(oc.mod)
R> predict(oc.mod, x_of, type=c("class","raw"), supplemental.cols=c("x","y"))
'2' '3' x y CLUSTER_ID
1 3.616386e-08 9.999999e-01 3.825303 3.935346 3
2 3.253662e-01 6.746338e-01 3.454143 4.193395 3
3 3.616386e-08 9.999999e-01 4.049120 4.172898 3
# ... Intervening rows not shown.
98 1.000000e+00 1.275712e-12 2.011463 1.991468 2
99 1.000000e+00 1.275712e-12 1.727580 1.898839 2
100 1.000000e+00 1.275712e-12 2.092737 2.212688 2
4.2.13 特異値分解
ore.odmSVD関数は、OML4SQLの特異値分解(SVD)アルゴリズムを使用するモデルを作成します。
特異値分解(SVD)は特徴抽出アルゴリズムです。SVDは、矩形行列を3つの行列('U'、'D'および'V')に分解することで、基礎となるデータの分散を取得する直交線形変換です。行列'D'は対角行列であり、その特異値は、ベースによって取得されるデータ分散の量を反映しています。
特異値分解モデルの設定
次の表に、単数値分解モデルに適用される設定を示します。
表4-16 特異値分解モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
2500 |
SVDによってサポートされている特徴の最大数。 |
例4-23 ore.odmSVD関数の使用方法
IRIS <- ore.push(cbind(Id = seq_along(iris[[1L]]), iris))
svd.mod <- ore.odmSVD(~. -Id, IRIS)
summary(svd.mod)
d(svd.mod)
v(svd.mod)
head(predict(svd.mod, IRIS, supplemental.cols = "Id"))
svd.pmod <- ore.odmSVD(~. -Id, IRIS,
odm.settings = list(odms_partition_columns = "Species"))
summary(svd.pmod)
d(svd.pmod)
v(svd.pmod)
head(predict(svd.pmod, IRIS, supplemental.cols = "Id"))
この例のリスト
R> IRIS <- ore.push(cbind(Id = seq_along(iris[[1L]]), iris))
R>
R> svd.mod <- ore.odmSVD(~. -Id, IRIS)
R> summary(svd.mod)
Call:
ore.odmSVD(formula = ~. - Id, data = IRIS)
Settings:
value
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto ON
scoring.mode scoring.svd
u.matrix.output u.matrix.disable
d:
FEATURE_ID VALUE
1 1 96.2182677
2 2 19.0780817
3 3 7.2270380
4 4 3.1502152
5 5 1.8849634
6 6 1.1474731
7 7 0.5814097
v:
ATTRIBUTE_NAME ATTRIBUTE_VALUE '1' '2' '3' '4' '5' '6' '7'
1 Petal.Length <NA> 0.51162932 0.65943465 -0.004420703 0.05479795 -0.51969015 0.17392232 -0.005674672
2 Petal.Width <NA> 0.16745698 0.32071102 0.146484369 0.46553390 0.72685033 0.31962337 -0.021274748
3 Sepal.Length <NA> 0.74909171 -0.26482593 -0.102057243 -0.49272847 0.31969417 -0.09379235 -0.067308615
4 Sepal.Width <NA> 0.37906736 -0.50824062 0.142810811 0.69139828 -0.25849391 -0.17606099 -0.041908520
5 Species setosa 0.03170407 -0.32247642 0.184499940 -0.12245506 -0.14348647 0.76017824 0.497502783
6 Species versicolor 0.04288799 0.04054823 -0.780684855 0.19827972 0.07363250 -0.12354271 0.571881302
7 Species virginica 0.05018593 0.16796988 0.551546107 -0.07177990 0.08109974 -0.48442099 0.647048040
Warning message:
In u.ore.odmSVD(object) : U matrix is not calculated.
R> d(svd.mod)
FEATURE_ID VALUE
1 1 96.2182677
2 2 19.0780817
3 3 7.2270380
4 4 3.1502152
5 5 1.8849634
6 6 1.1474731
7 7 0.5814097
Warning message:
ORE object has no unique key - using random order
R> v(svd.mod)
ATTRIBUTE_NAME ATTRIBUTE_VALUE '1' '2' '3' '4' '5' '6' '7'
1 Petal.Length <NA> 0.51162932 0.65943465 -0.004420703 0.05479795 -0.51969015 0.17392232 -0.005674672
2 Petal.Width <NA> 0.16745698 0.32071102 0.146484369 0.46553390 0.72685033 0.31962337 -0.021274748
3 Sepal.Length <NA> 0.74909171 -0.26482593 -0.102057243 -0.49272847 0.31969417 -0.09379235 -0.067308615
4 Sepal.Width <NA> 0.37906736 -0.50824062 0.142810811 0.69139828 -0.25849391 -0.17606099 -0.041908520
5 Species setosa 0.03170407 -0.32247642 0.184499940 -0.12245506 -0.14348647 0.76017824 0.497502783
6 Species versicolor 0.04288799 0.04054823 -0.780684855 0.19827972 0.07363250 -0.12354271 0.571881302
7 Species virginica 0.05018593 0.16796988 0.551546107 -0.07177990 0.08109974 -0.48442099 0.647048040
Warning message:
ORE object has no unique key - using random order
R> head(predict(svd.mod, IRIS, supplemental.cols = "Id"))
Id '1' '2' '3' '4' '5' '6' '7' FEATURE_ID
1 1 0.06161595 -0.1291839 0.02586865 -0.01449182 1.536727e-05 -0.023495349 -0.007998605 2
2 2 0.05808905 -0.1130876 0.01881265 -0.09294788 3.466226e-02 0.069569113 0.051195429 2
3 3 0.05678818 -0.1190959 0.02565027 -0.01950986 8.851560e-04 0.040073030 0.060908867 2
4 4 0.05667915 -0.1081308 0.02496402 -0.02233741 -5.750222e-02 0.093904181 0.077741713 2
5 5 0.06123138 -0.1304597 0.02925687 0.02309694 -3.065834e-02 -0.030664898 -0.003629897 2
6 6 0.06747071 -0.1302726 0.03340671 0.06114966 -9.547838e-03 -0.008210224 -0.081807741 2
R>
R> svd.pmod <- ore.odmSVD(~. -Id, IRIS,
+ odm.settings = list(odms_partition_columns = "Species"))
R> summary(svd.pmod)
$setosa
Call:
ore.odmSVD(formula = ~. - Id, data = IRIS, odm.settings = list(odms_partition_columns = "Species"))
Settings:
value
odms.max.partitions 1000
odms.missing.value.treatment odms.missing.value.auto
odms.partition.columns "Species"
odms.sampling odms.sampling.disable
prep.auto ON
scoring.mode scoring.svd
u.matrix.output u.matrix.disable
d:
FEATURE_ID VALUE
1 1 44.2872290
2 2 1.5719162
3 3 1.1458732
4 4 0.6836692
v:
ATTRIBUTE_NAME ATTRIBUTE_VALUE '1' '2' '3' '4'
1 Petal.Length <NA> 0.2334487 0.46456598 0.8317440 -0.19463332
2 Petal.Width <NA> 0.0395488 0.04182015 0.1946750 0.97917752
3 Sepal.Length <NA> 0.8010073 0.40303704 -0.4410167 0.03811461
4 Sepal.Width <NA> 0.5498408 -0.78739486 0.2753323 -0.04331888
$versicolor
Call:
ore.odmSVD(formula = ~. - Id, data = IRIS, odm.settings = list(odms_partition_columns = "Species"))
Settings:
value
odms.max.partitions 1000
odms.missing.value.treatment odms.missing.value.auto
R> # xyz
R> d(svd.pmod)
PARTITION_NAME FEATURE_ID VALUE
1 setosa 1 44.2872290
2 setosa 2 1.5719162
3 setosa 3 1.1458732
4 setosa 4 0.6836692
5 versicolor 1 56.2523412
6 versicolor 2 1.9106625
7 versicolor 3 1.7015929
8 versicolor 4 0.6986103
9 virginica 1 66.2734064
10 virginica 2 2.4318639
11 virginica 3 1.6007740
12 virginica 4 1.2958261
Warning message:
ORE object has no unique key - using random order
R> v(svd.pmod)
PARTITION_NAME ATTRIBUTE_NAME ATTRIBUTE_VALUE '1' '2' '3' '4'
1 setosa Petal.Length <NA> 0.2334487 0.46456598 0.83174398 -0.19463332
2 setosa Petal.Width <NA> 0.0395488 0.04182015 0.19467497 0.97917752
3 setosa Sepal.Length <NA> 0.8010073 0.40303704 -0.44101672 0.03811461
4 setosa Sepal.Width <NA> 0.5498408 -0.78739486 0.27533228 -0.04331888
5 versicolor Petal.Length <NA> 0.5380908 0.49576111 -0.60174021 -0.32029352
6 versicolor Petal.Width <NA> 0.1676394 0.36693207 -0.03448373 0.91436795
7 versicolor Sepal.Length <NA> 0.7486029 -0.64738491 0.06943054 0.12516311
8 versicolor Sepal.Width <NA> 0.3492119 0.44774385 0.79492074 -0.21372297
9 virginica Petal.Length <NA> 0.5948985 -0.26368708 0.65157671 -0.38988802
10 virginica Petal.Width <NA> 0.2164036 0.59106806 0.42921836 0.64774968
11 virginica Sepal.Length <NA> 0.7058813 -0.27846153 -0.53436210 0.37235450
12 virginica Sepal.Width <NA> 0.3177999 0.70962445 -0.32507927 -0.53829342
Warning message:
ORE object has no unique key - using random order
R> head(predict(svd.pmod, IRIS, supplemental.cols = "Id"))
Id '1' '2' '3' '4' FEATURE_ID
1 1 0.1432539 -0.026487881 -0.071688339 -0.04956008 1
2 2 0.1334289 0.172689424 -0.114854368 -0.02902893 2
3 3 0.1317675 -0.008327214 -0.062409295 -0.02438248 1
4 4 0.1297716 0.075232572 0.097222019 -0.08055912 1
5 5 0.1426868 -0.102219140 -0.009172782 -0.06147133 1
6 6 0.1554060 -0.055950655 0.160698708 0.14286095 34.2.14 サポート・ベクター・マシン
ore.odmSVM関数は、OML4Rのサポート・ベクター・マシン(SVM)モデルを構築します。
SVMは、Vapnik-Chervonenkis理論に基づいた強固な理論的基礎を持つ最新の強力なアルゴリズムです。SVMは、強力な正則化プロパティを持ちます。正則化とは、新しいデータへのモデルの一般化を指します。
SVMモデルは、一般的な機械学習手法であるニューラル・ネットワークおよび動径基底関数に似た関数形式を持ちます。
SVMは、次のような問題の解決に使用できます。
-
分類: SVM分類は、決定境界を定義する決定面の概念に基づいています。決定面は、異なるクラスのメンバーシップを持つオブジェクト・セット間を区別するものです。SVMは、クラスの最も広範な区切りを与えるセパレータを定義するベクター(サポート・ベクター)を検出します。
SVM分類では、2項および多クラスの両ターゲットがサポートされます。
-
回帰: SVMでは、回帰問題を解決するために、イプシロン非感受性損失関数が使用されます。
SVM回帰では、データ・ポイントの最大数がイプシロン幅の非感受性チューブ内に収まるような連続関数の検出が試行されます。真のターゲット値のイプシロン距離内におさまる予測は、誤差として解釈されません。
-
異常検出: 異常検出は、一見同質なデータ内に存在する特異なケースを識別します。異常検出は、重大な意味を持ちながら検出することが難しい、不正行為、ネットワークへの侵入などの発生頻度の低いイベントを検出するための重要なツールです。
異常検出は、1クラスSVM分類として実装されます。異常検出モデルでは、あるデータ・ポイントが特定の分布に対して典型的かどうかを予測します。
ore.odmSVM関数は、次の3つの異なるタイプのモデルを構築します。一部、分類モデルにのみ適用される引数、回帰モデルにのみ適用される引数、異常検出モデルにのみ適用される引数があります。
ore.odmSVM関数の引数の詳細は、help(ore.odmSVM)をコールしてください。
サポート・ベクター・マシン・モデルの設定
次の表に、Support Vector Machineモデルに適用される設定を示します。
表4-17 サポート・ベクター・マシン・モデルの設定
| 設定名 | 設定値 | 説明 |
|---|---|---|
|
|
|
新しいデータに対して優れた一般化を実現するための、モデルの堅牢性に対するモデルの複雑性のバランスをとる正則化設定。SVMでは、データ駆動の手法を使用して複雑性要素が検出される。 SVMアルゴリズム(分類と回帰の両方)の複雑度の値。 デフォルト値は、アルゴリズムによってデータから概算されます。 |
|
|
|
SVMアルゴリズムでの収束許容値。 デフォルトは |
|
|
|
回帰用の正則化設定(複雑性要素と似ている)。イプシロンによって、データ内で許容される残差(ノイズ)が指定される。 SVM回帰のイプシロン係数の値。 デフォルトは |
|
|
|
サポート・ベクター・マシンのカーネル。線形またはガウス。 デフォルト値は |
|
|
|
トレーニング・データでの目標となる外れ値率。1クラスSVMモデル(異常検出)に対してのみ有効です。 デフォルトは |
SVMS_STD_DEV |
|
ガウス・カーネル関数の分布を制御する。SVMでは、データ駆動の手法が使用され、通常のケース間の距離と同じスケールにある標準偏差値が検出される。 SVMアルゴリズムの標準偏差の値。 これは、ガウス・カーネルにのみ適用されます。 デフォルト値は、アルゴリズムによってデータから概算されます。 |
|
|
正の整数 |
この設定により、SVM反復数の上限が設定されます。これはSVMソルバーに依存するため、デフォルトはシステムによって決定されます。 |
SVMS_NUM_PIVOTS |
[ |
この設定により、不完全なコレスキ分解で使用されるピボット数の上限が設定されます。これは、非線形カーネルにのみ設定できます。デフォルト値は |
|
|
正の整数 |
この設定は、線形カーネルを使用するSVMモデルに適用されます。この設定により、SGDソルバーのバッチのサイズが設定されます。0を入力すると、データ駆動のバッチ・サイズの推定値がトリガーされます。デフォルトは |
|
|
|
この設定により、SGD SVMソルバーで使用される正則化のタイプが制御されます。この設定は、線形SVMモデルにのみ使用できます。これは潜在的なモデル・サイズに依存するため、デフォルトはシステムによって決定されます。 |
SVMS_SOLVER |
|
この設定により、ユーザーがSVMソルバーを選択できます。カーネルが非線形である場合、SGDソルバーは選択できません。デフォルト値は、システムによって決定されます。 |
例4-24 ore.odmSVM関数の使用方法および混同マトリクスの生成
この例では、SVM分類の使用方法を示します。この例では、Rのmtcarsデータセットからデータベースにmtcarsを作成し、分類モデルを構築し、予測を行い、最終的に、混同マトリクスを生成します。
m <- mtcars m$gear <- as.factor(m$gear) m$cyl <- as.factor(m$cyl) m$vs <- as.factor(m$vs) m$ID <- 1:nrow(m) mtcars_of <- ore.push(m) svm.mod <- ore.odmSVM(gear ~ .-ID, mtcars_of, "classification") summary(svm.mod) svm.res <- predict (svm.mod, mtcars_of,"gear") with(svm.res, table(gear, PREDICTION)) # generate confusion matrix
この例のリスト
R> m <- mtcars
R> m$gear <- as.factor(m$gear)
R> m$cyl <- as.factor(m$cyl)
R> m$vs <- as.factor(m$vs)
R> m$ID <- 1:nrow(m)
R> mtcars_of <- ore.push(m)
R>
R> svm.mod <- ore.odmSVM(gear ~ .-ID, mtcars_of, "classification")
R> summary(svm.mod)
Call:
ore.odmSVM(formula = gear ~ . - ID, data = mtcars_of, type = "classification")
Settings:
value
prep.auto on
active.learning al.enable
complexity.factor 0.385498
conv.tolerance 1e-04
kernel.cache.size 50000000
kernel.function gaussian
std.dev 1.072341
Coefficients:
[1] No coefficients with gaussian kernel
R> svm.res <- predict (svm.mod, mtcars_of,"gear")
R> with(svm.res, table(gear, PREDICTION)) # generate confusion matrix
PREDICTION
gear 3 4
3 12 3
4 0 12
5 2 3
例4-25 ore.odmSVM関数の使用方法および回帰モデルの構築
この例では、SVM回帰を示します。この例では、データ・フレームを作成し、それを表にプッシュしてから、回帰モデルを構築します(ore.odmSVMでは、線形カーネルを指定します)。
x <- seq(0.1, 5, by = 0.02) y <- log(x) + rnorm(x, sd = 0.2) dat <-ore.push(data.frame(x=x, y=y)) # Build model with linear kernel svm.mod <- ore.odmSVM(y~x,dat,"regression", kernel.function="linear") summary(svm.mod) coef(svm.mod) svm.res <- predict(svm.mod,dat, supplemental.cols="x") head(svm.res,6)
この例のリスト
R> x <- seq(0.1, 5, by = 0.02)
R> y <- log(x) + rnorm(x, sd = 0.2)
R> dat <-ore.push(data.frame(x=x, y=y))
R>
R> # Build model with linear kernel
R> svm.mod <- ore.odmSVM(y~x,dat,"regression", kernel.function="linear")
R> summary(svm.mod)
Call:
ore.odmSVM(formula = y ~ x, data = dat, type = "regression",
kernel.function = "linear")
Settings:
value
prep.auto on
active.learning al.enable
complexity.factor 0.620553
conv.tolerance 1e-04
epsilon 0.098558
kernel.function linear
Residuals:
Min. 1st Qu. Median Mean 3rd Qu. Max.
-0.79130 -0.28210 -0.05592 -0.01420 0.21460 1.58400
Coefficients:
variable value estimate
1 x 0.6637951
2 (Intercept) 0.3802170
R> coef(svm.mod)
variable value estimate
1 x 0.6637951
2 (Intercept) 0.3802170
R> svm.res <- predict(svm.mod,dat, supplemental.cols="x")
R> head(svm.res,6)
x PREDICTION
1 0.10 -0.7384312
2 0.12 -0.7271410
3 0.14 -0.7158507
4 0.16 -0.7045604
5 0.18 -0.6932702
6 0.20 -0.6819799
例4-26 ore.odmSVM関数の使用方法および異常検出モデルの構築
この例では、SVN異常検出を示します。ここでは、分類の例で作成されたmtcars_ofを使用し、異常検出モデルを構築します。
svm.mod <- ore.odmSVM(~ .-ID, mtcars_of, "anomaly.detection") summary(svm.mod) svm.res <- predict (svm.mod, mtcars_of, "ID") head(svm.res) table(svm.res$PREDICTION)
この例のリスト
R> svm.mod <- ore.odmSVM(~ .-ID, mtcars_of, "anomaly.detection")
R> summary(svm.mod)
Call:
ore.odmSVM(formula = ~. - ID, data = mtcars_of, type = "anomaly.detection")
Settings:
value
prep.auto on
active.learning al.enable
conv.tolerance 1e-04
kernel.cache.size 50000000
kernel.function gaussian
outlier.rate .1
std.dev 0.719126
Coefficients:
[1] No coefficients with gaussian kernel
R> svm.res <- predict (svm.mod, mtcars_of, "ID")
R> head(svm.res)
'0' '1' ID PREDICTION
Mazda RX4 0.4999405 0.5000595 1 1
Mazda RX4 Wag 0.4999794 0.5000206 2 1
Datsun 710 0.4999618 0.5000382 3 1
Hornet 4 Drive 0.4999819 0.5000181 4 1
Hornet Sportabout 0.4949872 0.5050128 5 1
Valiant 0.4999415 0.5000585 6 1
R> table(svm.res$PREDICTION)
0 1
5 27 4.2.15 パーティション化されたモデルの構築
パーティション化されたモデルは、複数のサブモデル(データの各パーティションに1つ)で構成されるアンサンブル・モデルです。
パーティション化されたモデルは、モデルとして管理および使用される複数のターゲット・モデルによって、精度を向上させることができます。パーティション化されたモデルでは、最上位のモデルのみを参照可能にして、スコアリングを簡素化できます。適切なサブモデルは、スコアリングの対象となるデータの各行に対するパーティション化された列の値に基づいて選択されます。
パーティション化されたOML4SQLモデルを作成するには、odm.setting引数を使用します。名前ODMS_PARTITION_COLUMNSと入力データを値としてパーティション化する列名を使用します。OREdm関数は、各パーティションのサブモデルとともにモデルを返します。パーティションは、列にある一意の値に基づいています。
partitions関数は、指定されたモデル・オブジェクトの各パーティションおよびモデルの関連付けられたパーティション列値をリストするore.frameを返します。パーティション名はシステムによって決定されます。関数は、パーティション化されていないモデルにNULLを返します。
例4-27 パーティション化されたモデルの作成
この例では、パーティション化されたサポート・ベクター・マシン分類モデルを作成します。ここでは、カリフォルニア大学アーバイン校機械学習リポジトリのワイン品質データセットを使用します。
# Download the wine data set and create the data table.
white.url <- "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-white.csv"
white.wine <- read.csv(white.url, header = TRUE, sep = ";")
white.wine$color <- "white"
red.url <- "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-red.csv"
red.wine <- read.csv(red.url, header = TRUE, sep = ";")
red.wine$color <- "red"
dat <- rbind(white.wine, red.wine)
# Drop the WINE table if it exists.
ore.drop(table="WINE")
ore.create(dat, table="WINE")
# Assign row names to enable row indexing for train and test samples.
row.names(WINE) <- WINE$color
# Enable reproducible results.
set.seed(seed=6218945)
n.rows <- nrow(WINE)
# Train and test sampling.
random.sample <- sample(1:n.rows, ceiling(n.rows/2))
# Sample in-database using row indexing.
WINE.train <- WINE[random.sample,]
WINE.test <- WINE[setdiff(1:n.rows,random.sample),]
# Build a Support Vector Machine classification model
# on the training data set, using both red and white wine.
mod.svm <- ore.odmSVM(quality~.-pH-fixed.acidity, WINE.train,
"classification", kernel.function="linear")
# Predict wine quality on the test data set.
pred.svm <- predict (mod.svm, WINE.test,"quality")
# View the probability of each class and prediction.
head(pred.svm,3)
# Generate a confusion matrix. Note that 3 and 8 are not predicted.
with(pred.svm, table(quality, PREDICTION, dnn = c("Actual", "Predicted")))
# Build a partitioned SVM model based on wine color.
# Specify the partitioning column with the odm.settings argument.
mod.svm2 <- ore.odmSVM(quality~.-pH-fixed.acidity, WINE.train,
"classification", kernel.function="linear",
odm.settings=list(odms_partition_columns = "color"))
# Predict wine quality on the test data set.
pred.svm2 <- predict (mod.svm2, WINE.test, "quality")
# View the probability of each class and prediction.
head(pred.svm2,3)
# Generate a confusion matrix. Note that 3 and 4 are not predicted.
with(pred.svm2, table(quality, PREDICTION, dnn = c("Actual", "Predicted")))
partitions(mod.svm2)
summary(mod.svm2["red"])この例のリスト
> # Download the wine data set and create the data table.
> white.url <- "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-white.csv"
> white.wine <- read.csv(white.url, header = TRUE, sep = ";")
> white.wine$color <- "white"
>
> red.url <- "https://archive.ics.uci.edu/ml/machine-learning-databases/wine-quality/winequality-red.csv"
> red.wine <- read.csv(red.url, header = TRUE, sep = ";")
> red.wine$color <- "red"
>
> dat <- rbind(white.wine, red.wine)
>
> # Drop the WINE table if it exists.
> ore.drop(table="WINE")
Warning message:
Table WINE does not exist.
> ore.create(dat, table="WINE")
>
> # Assign row names to enable row indexing for train and test samples.
> row.names(WINE) <- WINE$color
>
> # Enable reproducible results.
> set.seed(seed=6218945)
>
> n.rows <- nrow(WINE)
>
> # Train and test sampling.
> random.sample <- sample(1:n.rows, ceiling(n.rows/2))
>
> # Sample in-database using row indexing.
> WINE.train <- WINE[random.sample,]
> WINE.test <- WINE[setdiff(1:n.rows,random.sample),]
>
> # Build a Support Vector Machine classification model
> # on the training data set, using both red and white wine.
> mod.svm <- ore.odmSVM(quality~.-pH-fixed.acidity, WINE.train,
+ "classification",kernel.function="linear")
>
> # Predict wine quality on the test data set.
> pred.svm <- predict (mod.svm, WINE.test,"quality")
>
> # View the probability of each class and prediction.
> head(pred.svm,3)
'3' '4' '5' '6' '7' '8' '9'
red 0.04957242 0.1345280 0.27779399 0.1345281 0.1345280 0.1345275 0.1345220
red.1 0.04301663 0.1228311 0.34283345 0.1228313 0.1228311 0.1228307 0.1228257
red.2 0.04473419 0.1713883 0.09832961 0.1713891 0.1713890 0.1713886 0.1713812
quality PREDICTION
red 4 5
red.1 5 5
red.2 7 6
>
> # Generate a confusion matrix. Note that 3 and 4 are not predicted.
> with(pred.svm, table(quality,PREDICTION, dnn = c("Actual","Predicted")))
Predicted
Actual 3 4 5 6 7 8 9
3 0 0 11 5 0 0 0
4 0 1 85 16 2 0 0
5 2 1 927 152 4 0 1
6 2 1 779 555 63 1 9
7 2 0 121 316 81 0 3
8 0 0 18 66 21 0 0
9 0 0 0 2 1 0 0
>
> partitions(mod.svm2)
PARTITION_NAME color
1 red red
2 white white
> summary(mod.svm2["red"])
$red
Call:
ore.odmSVM(formula = quality ~ . - pH - fixed.acidity, data = WINE.train,
type = "classification", kernel.function = "linear", odm.settings = list(odms_partition_columns = "color"))
Settings:
value
clas.weights.balanced OFF
odms.details odms.enable
odms.max.partitions 1000
odms.missing.value.treatment odms.missing.value.auto
odms.partition.columns "color"
odms.sampling odms.sampling.disable
prep.auto ON
active.learning al.enable
conv.tolerance 1e-04
kernel.function linear
Coefficients:
PARTITION_NAME class variable value estimate
1 red 3 (Intercept) -1.347392e+01
2 red 3 alcohol 7.245737e-01
3 red 3 chlorides 1.761946e+00
4 red 3 citric.acid -3.276716e+00
5 red 3 density 2.449906e+00
6 red 3 free.sulfur.dioxide -6.035430e-01
7 red 3 residual.sugar 9.097631e-01
8 red 3 sulphates 1.240524e-04
9 red 3 total.sulfur.dioxide -2.467554e+00
10 red 3 volatile.acidity 1.300470e+00
11 red 4 (Intercept) -1.000002e+00
12 red 4 alcohol -7.920188e-07
13 red 4 chlorides -2.589198e-08
14 red 4 citric.acid 9.340296e-08
15 red 4 density -5.418190e-07
16 red 4 free.sulfur.dioxide -6.981268e-08
17 red 4 residual.sugar 3.389558e-07
18 red 4 sulphates 1.417324e-07
19 red 4 total.sulfur.dioxide -3.113900e-07
20 red 4 volatile.acidity 4.928625e-07
21 red 5 (Intercept) -3.151406e-01
22 red 5 alcohol -9.692192e-01
23 red 5 chlorides 3.690034e-02
24 red 5 citric.acid 2.258823e-01
25 red 5 density -1.770474e-01
26 red 5 free.sulfur.dioxide -1.289540e-01
27 red 5 residual.sugar 7.521771e-04
28 red 5 sulphates -3.596548e-01
29 red 5 total.sulfur.dioxide 5.688280e-01
30 red 5 volatile.acidity 3.005168e-01
31 red 6 (Intercept) -9.999994e-01
32 red 6 alcohol 8.807703e-07
33 red 6 chlorides 6.871310e-08
34 red 6 citric.acid -4.525750e-07
35 red 6 density 5.786923e-07
36 red 6 free.sulfur.dioxide 3.856018e-07
37 red 6 residual.sugar -4.281695e-07
38 red 6 sulphates 1.036468e-07
39 red 6 total.sulfur.dioxide -4.287512e-07
40 red 6 volatile.acidity -4.426151e-07
41 red 7 (Intercept) -1.000000e+00
42 red 7 alcohol 1.761665e-07
43 red 7 chlorides -3.583316e-08
44 red 7 citric.acid -4.837739e-08
45 red 7 density 2.169500e-08
46 red 7 free.sulfur.dioxide 4.800717e-08
47 red 7 residual.sugar 1.909498e-08
48 red 7 sulphates 1.062205e-07
49 red 7 total.sulfur.dioxide -2.339108e-07
50 red 7 volatile.acidity -1.539326e-07
51 red 8 (Intercept) -1.000000e+00
52 red 8 alcohol 7.089889e-08
53 red 8 chlorides -8.566726e-09
54 red 8 citric.acid 2.769301e-08
55 red 8 density -3.852321e-08
56 red 8 free.sulfur.dioxide -1.302056e-08
57 red 8 residual.sugar 4.847947e-09
58 red 8 sulphates 1.276461e-08
59 red 8 total.sulfur.dioxide -5.484427e-08
60 red 8 volatile.acidity 2.959182e-08
4.2.16 テキスト処理モデルの構築
テキスト処理モデルでは、ctx.settings引数を使用してOracle Text属性の設定を指定します。
例4-28 テキスト処理モデルの構築
この例では、テキストを処理するore.odmKMeansモデルを構築します。ここでは、odm.settings引数およびctx.settings引数を使用します。例の後の図に、histogram(km.mod1)関数の出力を示します。
x <- rbind(matrix(rnorm(100, sd = 0.3), ncol = 2),
matrix(rnorm(100, mean = 1, sd = 0.3), ncol = 2))
colnames(x) <- c("x", "y")
X <- ore.push (data.frame(x))
km.mod1 <- NULL
km.mod1 <- ore.odmKMeans(~., X, num.centers = 2)
km.mod1
summary(km.mod1)
rules(km.mod1)
clusterhists(km.mod1)
histogram(km.mod1)
km.res1 <- predict(km.mod1,X,type="class",supplemental.cols=c("x","y"))
head(km.res1,3)
km.res1.local <- ore.pull(km.res1)
plot(data.frame(x = km.res1.local$x,
y = km.res1.local$y),
col = km.res1.local$CLUSTER_ID)
points(km.mod1$centers2, col = rownames(km.mod1$centers2), pch = 8, cex=2)
head(predict(km.mod1,X))
head(predict(km.mod1,X,type=c("class","raw"),supplemental.cols=c("x","y")),3)
head(predict(km.mod1,X,type="raw",supplemental.cols=c("x","y")),3)
# Text processing with ore.odmKMeans.
title <- c('Aids in Africa: Planning for a long war',
'Mars rover maneuvers for rim shot',
'Mars express confirms presence of water at Mars south pole',
'NASA announces major Mars rover finding',
'Drug access, Asia threat in focus at AIDS summit',
'NASA Mars Odyssey THEMIS image: typical crater',
'Road blocks for Aids')
response <- c('Aids', 'Mars', 'Mars', 'Mars', 'Aids', 'Mars', 'Aids')
# Text contents in a character column.
KM_TEXT <- ore.push(data.frame(CUST_ID = seq(length(title)),
RESPONSE = response, TITLE = title))
# Create a text policy (CTXSYS.CTX_DDL privilege is required).
ore.exec("Begin ctx_ddl.create_policy('ESA_TXTPOL'); End;")
# Specify POLICY_NAME, MIN_DOCUMENTS, MAX_FEATURES and
# text column attributes.
km.mod <- ore.odmKMeans( ~ TITLE, data = KM_TEXT, num.centers = 2L,
odm.settings = list(ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL",
ODMS_TEXT_MIN_DOCUMENTS = 1,
ODMS_TEXT_MAX_FEATURES = 3,
kmns_distance = "dbms_data_mining.kmns_cosine",
kmns_details = "kmns_details_all"),
ctx.settings = list(TITLE = "TEXT(TOKEN_TYPE:STEM)"))
summary(km.mod)
settings(km.mod)
print(predict(km.mod, KM_TEXT, supplemental.cols = "RESPONSE"), digits = 3L)
ore.exec("Begin ctx_ddl.drop_policy('ESA_TXTPOL'); End;")この例のリスト
R> x <- rbind(matrix(rnorm(100, sd = 0.3), ncol = 2),
+ matrix(rnorm(100, mean = 1, sd = 0.3), ncol = 2))
R> colnames(x) <- c("x", "y")
R>
R> X <- ore.push (data.frame(x))
R> km.mod1 <- NULL
R> km.mod1 <- ore.odmKMeans(~., X, num.centers = 2)
R> km.mod1
Call:
ore.odmKMeans(formula = ~., data = X, num.centers = 2)
Settings:
value
clus.num.clusters 2
block.growth 2
conv.tolerance 0.01
details details.all
distance euclidean
iterations 3
min.pct.attr.support 0.1
num.bins 10
random.seed 0
split.criterion variance
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto ON
R> summary(km.mod1)
Call:
ore.odmKMeans(formula = ~., data = X, num.centers = 2)
Settings:
value
clus.num.clusters 2
block.growth 2
conv.tolerance 0.01
details details.all
distance euclidean
iterations 3
min.pct.attr.support 0.1
num.bins 10
random.seed 0
split.criterion variance
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
prep.auto ON
Centers:
x y
2 -0.07638266 0.04449368
3 0.98493306 1.00864399
R> rules(km.mod1)
cluster.id rhs.support rhs.conf lhr.support lhs.conf lhs.var lhs.var.support lhs.var.conf predicate
1 1 100 1.0 92 0.86 x 86 0.2222222 x <= 1.2209
2 1 100 1.0 92 0.86 x 86 0.2222222 x >= -.6188
3 1 100 1.0 86 0.86 y 86 0.4444444 y <= 1.1653
4 1 100 1.0 86 0.86 y 86 0.4444444 y > -.3053
5 2 50 0.5 48 0.96 x 48 0.0870793 x <= .4324
6 2 50 0.5 48 0.96 x 48 0.0870793 x >= -.6188
7 2 50 0.5 48 0.96 y 48 0.0893300 y <= .5771
8 2 50 0.5 48 0.96 y 48 0.0893300 y > -.5995
9 3 50 0.5 49 0.98 x 49 0.0852841 x <= 1.7465
10 3 50 0.5 49 0.98 x 49 0.0852841 x > .4324
11 3 50 0.5 50 0.98 y 49 0.0838225 y <= 1.7536
12 3 50 0.5 50 0.98 y 49 0.0838225 y > .2829
R> clusterhists(km.mod1)
cluster.id variable bin.id lower.bound upper.bound label count
1 1 x 1 -0.61884662 -0.35602715 -.6188466:-.3560272 6
2 1 x 2 -0.35602715 -0.09320769 -.3560272:-.0932077 17
3 1 x 3 -0.09320769 0.16961178 -.0932077:.1696118 15
4 1 x 4 0.16961178 0.43243125 .1696118:.4324312 11
5 1 x 5 0.43243125 0.69525071 .4324312:.6952507 8
6 1 x 6 0.69525071 0.95807018 .6952507:.9580702 17
7 1 x 7 0.95807018 1.22088965 .9580702:1.2208896 18
8 1 x 8 1.22088965 1.48370911 1.2208896:1.4837091 4
9 1 x 9 1.48370911 1.74652858 1.4837091:1.7465286 4
10 1 y 1 -0.89359597 -0.59946141 -.893596:-.5994614 2
11 1 y 2 -0.59946141 -0.30532685 -.5994614:-.3053269 4
12 1 y 3 -0.30532685 -0.01119230 -.3053269:-.0111923 11
13 1 y 4 -0.01119230 0.28294226 -.0111923:.2829423 24
14 1 y 5 0.28294226 0.57707682 .2829423:.5770768 13
15 1 y 6 0.57707682 0.87121138 .5770768:.8712114 12
16 1 y 7 0.87121138 1.16534593 .8712114:1.1653459 26
17 1 y 8 1.16534593 1.45948049 1.1653459:1.4594805 5
18 1 y 9 1.45948049 1.75361505 1.4594805:1.753615 3
19 2 x 1 -0.61884662 -0.35602715 -.6188466:-.3560272 6
20 2 x 2 -0.35602715 -0.09320769 -.3560272:-.0932077 17
21 2 x 3 -0.09320769 0.16961178 -.0932077:.1696118 15
22 2 x 4 0.16961178 0.43243125 .1696118:.4324312 10
23 2 x 5 0.43243125 0.69525071 .4324312:.6952507 2
24 2 x 6 0.69525071 0.95807018 .6952507:.9580702 0
25 2 x 7 0.95807018 1.22088965 .9580702:1.2208896 0
26 2 x 8 1.22088965 1.48370911 1.2208896:1.4837091 0
27 2 x 9 1.48370911 1.74652858 1.4837091:1.7465286 0
28 2 y 1 -0.89359597 -0.59946141 -.893596:-.5994614 2
29 2 y 2 -0.59946141 -0.30532685 -.5994614:-.3053269 4
30 2 y 3 -0.30532685 -0.01119230 -.3053269:-.0111923 11
31 2 y 4 -0.01119230 0.28294226 -.0111923:.2829423 24
32 2 y 5 0.28294226 0.57707682 .2829423:.5770768 9
33 2 y 6 0.57707682 0.87121138 .5770768:.8712114 0
34 2 y 7 0.87121138 1.16534593 .8712114:1.1653459 0
35 2 y 8 1.16534593 1.45948049 1.1653459:1.4594805 0
36 2 y 9 1.45948049 1.75361505 1.4594805:1.753615 0
37 3 x 1 -0.61884662 -0.35602715 -.6188466:-.3560272 0
38 3 x 2 -0.35602715 -0.09320769 -.3560272:-.0932077 0
39 3 x 3 -0.09320769 0.16961178 -.0932077:.1696118 0
40 3 x 4 0.16961178 0.43243125 .1696118:.4324312 1
41 3 x 5 0.43243125 0.69525071 .4324312:.6952507 6
42 3 x 6 0.69525071 0.95807018 .6952507:.9580702 17
43 3 x 7 0.95807018 1.22088965 .9580702:1.2208896 18
44 3 x 8 1.22088965 1.48370911 1.2208896:1.4837091 4
45 3 x 9 1.48370911 1.74652858 1.4837091:1.7465286 4
46 3 y 1 -0.89359597 -0.59946141 -.893596:-.5994614 0
47 3 y 2 -0.59946141 -0.30532685 -.5994614:-.3053269 0
48 3 y 3 -0.30532685 -0.01119230 -.3053269:-.0111923 0
49 3 y 4 -0.01119230 0.28294226 -.0111923:.2829423 0
50 3 y 5 0.28294226 0.57707682 .2829423:.5770768 4
51 3 y 6 0.57707682 0.87121138 .5770768:.8712114 12
52 3 y 7 0.87121138 1.16534593 .8712114:1.1653459 26
53 3 y 8 1.16534593 1.45948049 1.1653459:1.4594805 5
54 3 y 9 1.45948049 1.75361505 1.4594805:1.753615 3
R> histogram(km.mod1)
R>
R> km.res1 <- predict(km.mod1, X, type="class", supplemental.cols = c("x","y"))
R> head(km.res1, 3)
x y CLUSTER_ID
1 -0.43646407 0.26201831 2
2 -0.02797831 0.07319952 2
3 0.11998373 -0.08638716 2
R> km.res1.local <- ore.pull(km.res1)
R> plot(data.frame(x = km.res1.local$x,
+ y = km.res1.local$y),
+ col = km.res1.local$CLUSTER_ID)
R> points(km.mod1$centers2, col = rownames(km.mod1$centers2), pch = 8, cex = 2)
R>
R> head(predict(km.mod1, X))
'2' '3' CLUSTER_ID
1 0.9992236 0.0007763706 2
2 0.9971310 0.0028690375 2
3 0.9974216 0.0025783939 2
4 0.9997335 0.0002665114 2
5 0.9917773 0.0082226599 2
6 0.9771667 0.0228333398 2
R> head(predict(km.mod1,X,type=c("class","raw"),supplemental.cols=c("x","y")),3)
'2' '3' x y CLUSTER_ID
1 0.9992236 0.0007763706 -0.43646407 0.26201831 2
2 0.9971310 0.0028690375 -0.02797831 0.07319952 2
3 0.9974216 0.0025783939 0.11998373 -0.08638716 2
R> head(predict(km.mod1,X,type="raw",supplemental.cols=c("x","y")),3)
x y '2' '3'
1 -0.43646407 0.26201831 0.9992236 0.0007763706
2 -0.02797831 0.07319952 0.9971310 0.0028690375
3 0.11998373 -0.08638716 0.9974216 0.0025783939R>
R>
R> # Text processing with ore.odmKMeans.
R> title <- c('Aids in Africa: Planning for a long war',
+ 'Mars rover maneuvers for rim shot',
+ 'Mars express confirms presence of water at Mars south pole',
+ 'NASA announces major Mars rover finding',
+ 'Drug access, Asia threat in focus at AIDS summit',
+ 'NASA Mars Odyssey THEMIS image: typical crater',
+ 'Road blocks for Aids')
R> response <- c('Aids', 'Mars', 'Mars', 'Mars', 'Aids', 'Mars', 'Aids')
R>
R> # Text contents in a character column.
R> KM_TEXT <- ore.push(data.frame(CUST_ID = seq(length(title)),
+ RESPONSE = response, TITLE = title))
R>
R> # Create a text policy (CTXSYS.CTX_DDL privilege is required).
R> ore.exec("Begin ctx_ddl.create_policy('ESA_TXTPOL'); End;")
R>
R> # Specify POLICY_NAME, MIN_DOCUMENTS, MAX_FEATURES and
R> # text column attributes.
R> km.mod <- ore.odmKMeans( ~ TITLE, data = KM_TEXT, num.centers = 2L,
+ odm.settings = list(ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL",
+ ODMS_TEXT_MIN_DOCUMENTS = 1,
+ ODMS_TEXT_MAX_FEATURES = 3,
+ kmns_distance = "dbms_data_mining.kmns_cosine",
+ kmns_details = "kmns_details_all"),
+ ctx.settings = list(TITLE="TEXT(TOKEN_TYPE:STEM)"))
R> summary(km.mod)
Call:
ore.odmKMeans(formula = ~TITLE, data = KM_TEXT, num.centers = 2L,
odm.settings = list(ODMS_TEXT_POLICY_NAME = "ESA_TXTPOL",
ODMS_TEXT_MIN_DOCUMENTS = 1, ODMS_TEXT_MAX_FEATURES = 3,
kmns_distance = "dbms_data_mining.kmns_cosine",
kmns_details = "kmns_details_all"),
ctx.settings = list(TITLE = "TEXT(TOKEN_TYPE:STEM)"))
Settings:
value
clus.num.clusters 2
block.growth 2
conv.tolerance 0.01
details details.all
distance cosine
iterations 3
min.pct.attr.support 0.1
num.bins 10
random.seed 0
split.criterion variance
odms.missing.value.treatment odms.missing.value.auto
odms.sampling odms.sampling.disable
odms.text.max.features 3
odms.text.min.documents 1
odms.text.policy.name ESA_TXTPOL
prep.auto ON
Centers:
TITLE.MARS TITLE.NASA TITLE.ROVER TITLE.AIDS
2 0.5292307 0.7936566 0.7936566 NA
3 NA NA NA 1
R> settings(km.mod)
SETTING_NAME SETTING_VALUE SETTING_TYPE
1 ALGO_NAME ALGO_KMEANS INPUT
2 CLUS_NUM_CLUSTERS 2 INPUT
3 KMNS_BLOCK_GROWTH 2 INPUT
4 KMNS_CONV_TOLERANCE 0.01 INPUT
5 KMNS_DETAILS KMNS_DETAILS_ALL INPUT
6 KMNS_DISTANCE KMNS_COSINE INPUT
7 KMNS_ITERATIONS 3 INPUT
8 KMNS_MIN_PCT_ATTR_SUPPORT 0.1 INPUT
9 KMNS_NUM_BINS 10 INPUT
10 KMNS_RANDOM_SEED 0 DEFAULT
11 KMNS_SPLIT_CRITERION KMNS_VARIANCE INPUT
12 ODMS_MISSING_VALUE_TREATMENT ODMS_MISSING_VALUE_AUTO DEFAULT
13 ODMS_SAMPLING ODMS_SAMPLING_DISABLE DEFAULT
14 ODMS_TEXT_MAX_FEATURES 3 INPUT
15 ODMS_TEXT_MIN_DOCUMENTS 1 INPUT
16 ODMS_TEXT_POLICY_NAME ESA_TXTPOL INPUT
17 PREP_AUTO ON INPUT
R> print(predict(km.mod, KM_TEXT, supplemental.cols = "RESPONSE"), digits = 3L)
'2' '3' RESPONSE CLUSTER_ID
1 0.0213 0.9787 Aids 3
2 0.9463 0.0537 Mars 2
3 0.9325 0.0675 Mars 2
4 0.9691 0.0309 Mars 2
5 0.0213 0.9787 Aids 3
6 0.9463 0.0537 Mars 2
7 0.0213 0.9787 Aids 3
R>
R> ore.exec("Begin ctx_ddl.drop_policy('ESA_TXTPOL'); End;")4.3 交差検定モデル
交差検定とはモデル改善手法の1つであり、使用可能なデータから繰り返しサンプリングを行って複数のモデルを構築およびテストすることにより、1回の学習/試験実験に伴う制約を回避します。
予測モデルは通常、特定のデータに基づいて構築され、別に保存されているデータや未知データに基づいて検証されます。交差検定の目的は、モデルが新しいデータに対して適切に一般化されるかを見極め、過剰適合の発生や、誤解を招きやすい既知データ特徴から誤った結論が導き出されるのを回避することにあります。
ore.CVユーティリティのR関数は、Oracle Machine Learning for Rを使用して回帰モデルと分類モデルの交差検定を実行します。
関数ore.CVは、アルゴリズムおよびケースの選択セットに対して、OML4Rの回帰関数および分類関数によりインデータベース・データを使用して生成されたモデルに対する交差検定を実行します。
ore.CV関数は、次のOML4R関数により生成されたモデルに使用できます。
-
ore.lm -
ore.stepwise -
ore.glm -
ore.neural -
ore.odmDT -
ore.odmGLM -
ore.odmNB -
ore.odmSVM
また、ore.CVを使用すると、OML4R埋込みRの実行によってR回帰関数で生成されたモデルを交差検定することもできます。これらのR関数は次のとおりです。
-
lm -
glm -
svm
関数ore.CVをダウンロードする方法、およびore.CVの使用方法の詳細と使用例は、次のブログ投稿を参照してください。