1.3 埋込みRの実行
埋込みRの実行を使用すると、ユーザーは、データベース環境によって生成されたRエンジンを使用してユーザー定義関数(UDF)のR関数を実行できます。この機能により、R関数へのデータベース・データのロード処理を自動化し、構造化された結果と構造化されていない結果(イメージを含む)の両方を返すことができます。ユーザーは、データベース・スクリプト・リポジトリにR関数を格納して管理できます。
埋込みRの実行:
Oracle DatabaseとOracle Autonomous Databaseの両方が、Rで記述されたカスタム関数である埋込みRの実行のUDFの作成とデプロイメントをサポートしています。これらの関数は、RおよびSQL問合せから直接呼び出すことができます。さらに、Oracle Autonomous Databaseは、この機能をREST APIに拡張し、外部アプリケーションおよびワークフローとのより優れた統合オプションを提供します。
ノート:
Oracle Databaseでは、Oracle REST Data Services (ORDS)を使用してREST APIを活用できるため、データベース内モデルへのWebベースのアクセスが可能になります。サードパーティ・パッケージ:
Oracle DatabaseとOracle Autonomous Databaseの両方で、ユーザーはR、またはRおよびSQLインタフェースからコールされるUDFで、サードパーティ・パッケージを活用できます。Oracle Databaseでは、Rのネイティブ・パッケージ管理ツールを使用してサード・パーティ・パッケージにアクセスできます。Oracle Autonomous Databaseは、Conda環境を介してサードパーティ・パッケージへのアクセスを提供し、この機能をREST APIに拡張して、機械学習ワークフローの柔軟性を高めます。
デプロイメントと使用
埋込みR実行により、本番環境でのユーザー定義R関数のデプロイメントが容易になります。これらの関数は、SQLインタフェースを使用して実行でき、Autonomous Databaseではアプリケーションへのシームレスな統合のためにRESTインタフェースを介して実行できます。
Oracle Databaseでは、並列処理用に3つの主要なタイプの関数がサポートされています:
- 非並列呼出し:
- doEval: 単一のRエンジンで関数を実行し、data.framesおよびイメージを含む結果を返します。表または結果セットの各行に対してユーザー定義関数を実行します。
- tableApply: 単一のRエンジンで関数を実行し、プロキシ・オブジェクトによって参照されるデータベース・データをRのdata.frameとして渡します。表または結果セット全体にユーザー定義関数を適用します。
- データの並列性:
- rowApply: 複数のRエンジンが使用される可能性のある行のチャンクでデータを処理することで、データ並列性を実装します。表または結果セットの各行にユーザー定義関数を並列で適用します。
- groupApply: 指定した列でデータベース・データをパーティション化し、複数のRエンジンが使用される可能性のある各パーティションにユーザー定義R関数を適用することで、データ並列性を有効にします。表または結果セットの各行のグループにユーザー定義関数を並列で適用します。
- タスクの並列性:
- indexApply: 複数のRエンジンが使用される可能性のある様々な索引値を使用したユーザー定義のR関数を複数回実行することで、タスクの並列化を促進します。ユーザー定義関数を索引の範囲に並列で適用します。
ユーザー定義関数(UDF)内でデータベース表またはビューを操作するには、透過層を利用します。この機能は、ore.doEvalやore.indexApplyなどの埋込み実行APIが表やビューをRエンジンに直接ロードしない場合に不可欠です。このような場合、R環境とデータベース間でデータのやり取りが透過的に発生します。透過層の範囲外のUDF操作の場合、計算はRエンジン内で実行されます。逆に、ore.tableApply、ore.groupApply、ore.rowApplyなどの関数は、インメモリー処理のためにデータをデータ・フレームとしてデータベースからRエンジンに明示的に転送します。
Rエンジンに表またはビューを直接ロードしないUDFの場合、透過層によってR環境とデータベース間のシームレスなデータ転送が保証されます。計算は、透過層の機能の外側でUDF操作のRエンジン内で実行されます。ore.tableApply、ore.groupApply、ore.rowApplyなどの埋込みR実行関数は、インメモリー処理のためにデータをデータ・フレームとしてデータベースからRエンジンに明示的に転送します。
例: 埋込みR UDFでの透過層の使用
ore.doEval(function(){
test <- ore.pull("TEST") # transparency layer
dim(test) # R
}
ore.doEval(function(){
test <- ore.sync("TEST") # transparency layer
ore.attach(test) # transparency layer
dim(test) # transparency layer, proxy object
}コードtest <- ore.pull("TEST")は、透過層関数ore.pullを使用して、TESTという名前のデータベース表からデータを取得します。その後、フェッチされたデータは変数testに格納されます。
埋込みRの実行の使用
これらの機能を使用するために、ユーザー定義のR関数がデータベース・スクリプト・リポジトリに格納されます。Autonomous Databaseでは、明示的な接続ステップを必要とせずに、接続が自動的に確立されます。
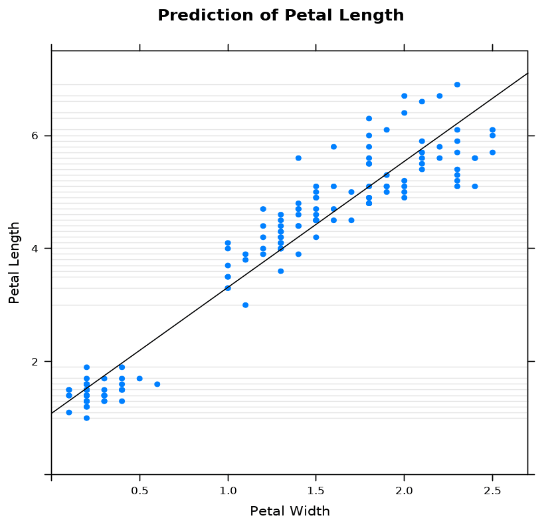
例1-1 xyplotを使用した線形モデルの作成
この例は、buildLM.1関数でlatticeライブラリのxyplotを使用して散布図を作成しています。
buildLM.1 <- function(dat){
library(lattice)
regr <- lm(Petal.Length~Petal.Width, dat)
x <- dat[['Petal.Width']]
y <- dat[['Petal.Length']]
print(xyplot(y~x, xlab = "Petal Width", ylab = "Petal Length",
panel = function(x,y) {
panel.dotplot(x,y)
panel.abline(lm(y ~ x))
},
xlim=c(0,2.7),
ylim=c(0,7.5),
main="Prediction of Petal Length"))
return(regr)
}%r buildLM.1(iris_df)例のリスト
Call:
lm(formula = Petal.Length ~ Petal.Width, data = dat)
Coefficients:
(Intercept) Petal.Width
1.084 2.230tableApply関数は、プロキシ・オブジェクトIRISを入力データとして受け入れ、ユーザー定義関数にデータ・フレームとして渡します。ユーザー定義関数は文字列として指定します。モデルがOMLオブジェクトとして返され、線形モデルであることを確認します。
%r
buildLM.1 <- function(dat,dsname){
library(lattice)
regr <- lm(Petal.Length~Petal.Width, dat)
x <- dat[['Petal.Width']]
y <- dat[['Petal.Length']]
xyplot(y~x, xlab = "Petal Width", ylab = "Petal Length",
panel = function(x,y) {
panel.dotplot(x,y)
panel.abline(lm(y ~ x))
},
xlim=c(0,2.7),
ylim=c(0,7.5),
main="Prediction of Petal Length")
return(regr)
}%r
mod <- ore.tableApply(IRIS,
buildLM.1,
dsname = 'ds-00')
cat("Type :",class(mod))
cat("\n")
cat("Coefficient :",(mod$coefficient))例のリストType : lm Coefficient : 1.083558 2.22994
このユーザー定義関数をスクリプト・リポジトリに保存し、関数名を指定して後で実行できるようになりました。
%r
ore.scriptCreate(name = 'buildLM.1',
FUN = buildLM.1,
overwrite = TRUE)
ore.scriptList(name = "buildLM.1")例のリストA data.frame: 1 x 2
NAME SCRIPT
<chr> <chr>
buildLM.1 function (dat)
{
library(lattice)
regr <- lm(Petal.Length ~ Petal.Width, dat)
x <- dat[["Petal.Width"]]
y <- dat[["Petal.Length"]]
print(xyplot(y ~ x, xlab = "Petal Width", ylab = "Petal Length", panel = function(x, y) {
panel.dotplot(x, y)
panel.abline(lm(y ~ x))
}, xlim = c(0, 2.7), ylim = c(0, 7.5), main = "Prediction of Petal Length"))
return(regr)
}